Visualizar anomalías de precipitación mensual
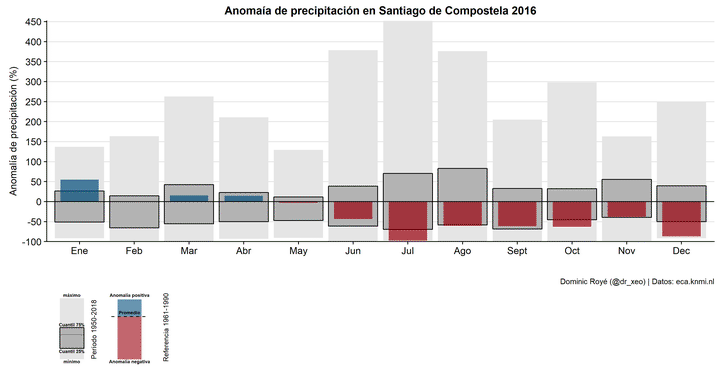
Normalmente cuando visualizamos anomalías de precipitación mensual, simplemente usamos un gráfico de barras indicando con color rojo y azul valores negativos y positivos. No obstante, no nos explica el contexto general de estas mismas anomalías. Por ejemplo, ¿cuál fue la anomalía más alta o más baja en cada mes? En principio, podríamos usar un boxplot para visualizar la distribución de las anomalías, pero en este caso concreto no encajarían bien estéticamente, por lo que debemos buscar una alternativa. Aquí os presento una forma gráfica muy útil.
Paquetes
En este post usaremos los siguientes paquetes:
| Paquete | Descripción |
|---|---|
| tidyverse | Conjunto de paquetes (visualización y manipulación de datos): ggplot2, dplyr, purrr,etc. |
| readr | Importar datos |
| ggthemes | Estilos para ggplot2 |
| lubridate | Fácil manipulación de fechas y tiempos |
| cowplot | Fácil creación de múltiples gráficos con ggplot2 |
#instalamos los paquetes si hace falta
if(!require("tidyverse")) install.packages("tidyverse")
if(!require("ggthemes")) install.packages("broom")
if(!require("cowplot")) install.packages("cowplot")
if(!require("lubridate")) install.packages("lubridate")
#paquetes
library(tidyverse) #contiene readr
library(ggthemes)
library(cowplot)
library(lubridate)Preparar los datos
Primero importamos la precipitación diaria de la estación meteorológica seleccionada (descarga). Usaremos datos de Santiago de Compostela (España) accesible a través de ECA&D.
Paso 1: importar los datos
No sólo importamos los datos en formato csv, sino también hacemos los primeros cambios. Saltamos las primeras 21 filas que contienen información sobre la estación meteorológica. Además, convertimos la fecha a clase date y reemplazamos valores ausentes (-9999) por NA. La precipitación está en 0.1 mm, por tanto, debemos dividir los valores por 10. Después seleccionamos las columnas DATE y RR, y las renombramos.
data <- read_csv("RR_STAID001394.txt", skip = 21) %>%
mutate(DATE = ymd(DATE), RR = ifelse(RR == -9999, NA, RR/10)) %>%
select(DATE:RR) %>%
rename(date = DATE, pr = RR)## Rows: 27606 Columns: 5## -- Column specification --------------------------------------------------------
## Delimiter: ","
## dbl (5): STAID, SOUID, DATE, RR, Q_RR##
## i Use `spec()` to retrieve the full column specification for this data.
## i Specify the column types or set `show_col_types = FALSE` to quiet this message.data## # A tibble: 27,606 x 2
## date pr
## <date> <dbl>
## 1 1943-11-01 0.6
## 2 1943-11-02 0
## 3 1943-11-03 0
## 4 1943-11-04 0
## 5 1943-11-05 0
## 6 1943-11-06 0
## 7 1943-11-07 0
## 8 1943-11-08 0
## 9 1943-11-09 0
## 10 1943-11-10 0
## # ... with 27,596 more rowsPaso 3: estimar las anomalías
Ahora debemos estimar los valores normales de cada mes y unir esta tabla a nuestros datos principales para posteriormente poder calcular la anomalía mensual. Expresamos la anomalía en porcentaje y restamos 100 para fijar el promedio en 0. Además, creamos una variable que nos indica si la anomalía es negativa o positiva, y otra con la fecha.
pr_ref <- filter(data, yr > 1981, yr <= 2010) %>%
group_by(mo) %>%
summarise(pr_ref = mean(prs))
data <- left_join(data, pr_ref, by = "mo")
data <- mutate(data,
anom = (prs*100/pr_ref)-100,
date = str_c(yr, as.numeric(mo), 1, sep = "-") %>% ymd(),
sign= ifelse(anom > 0, "pos", "neg") %>% factor(c("pos", "neg")))Ya podemos hacer un primer ensayo de un gráfico de anomalías (la versión clásica), para ello filtramos el año 2018. En este caso usamos la geometría de barras, recuerda que por defecto la función geom_bar() aplica el conteo a la variable. No obstante, en este caso conocemos y, por tanto indicamos con el argumento stat = "identity" que debe usar el valor dado en aes().
filter(data, yr == 2018) %>%
ggplot(aes(date, anom, fill = sign)) +
geom_bar(stat = "identity", show.legend = FALSE) +
scale_x_date(date_breaks = "month", date_labels = "%b") +
scale_y_continuous(breaks = seq(-100, 100, 20)) +
scale_fill_manual(values = c("#99000d", "#034e7b")) +
labs(y = "Anomalía de precipitación (%)", x = "") +
theme_hc()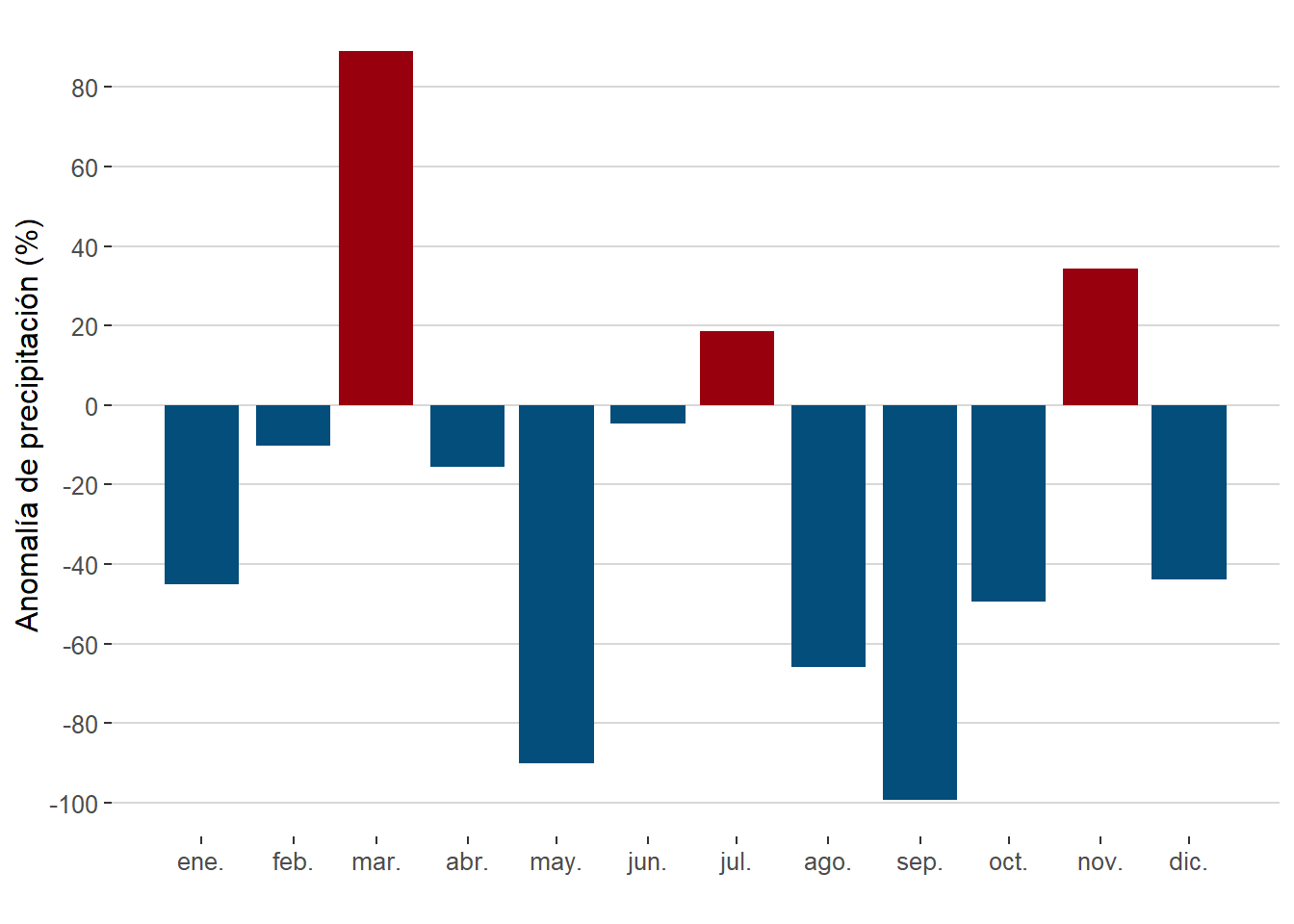
Paso 4: calcular las medidas estadísticas
En este último paso estimamos el valor máximo, mínimo, el cuantil 25%/75% y el rango intercuartil por mes de toda la serie temporal.
data_norm <- group_by(data, mo) %>%
summarise(mx = max(anom),
min = min(anom),
q25 = quantile(anom, .25),
q75 = quantile(anom, .75),
iqr = q75-q25)
data_norm## # A tibble: 12 x 6
## mo mx min q25 q75 iqr
## <ord> <dbl> <dbl> <dbl> <dbl> <dbl>
## 1 ene 193. -89.6 -43.6 56.3 99.9
## 2 feb 320. -96.5 -51.2 77.7 129.
## 3 mar 381. -100 -40.6 88.2 129.
## 4 abr 198. -93.6 -51.2 17.1 68.3
## 5 may 141. -90.1 -45.2 17.0 62.2
## 6 jun 419. -99.3 -58.2 50.0 108.
## 7 jul 311. -98.2 -77.3 27.1 104.
## 8 ago 264. -100 -68.2 39.8 108.
## 9 sep 241. -99.2 -64.9 48.6 113.
## 10 oct 220. -99.0 -54.5 4.69 59.2
## 11 nov 137. -98.8 -44.0 39.7 83.7
## 12 dic 245. -91.8 -49.8 36.0 85.8Crear el gráfico
Para crear el gráfico de anomalías con leyenda es necesario separar el gráfico principal de las leyendas.
Parte 1
En esta primera parte vamos añadiendo capa por capa los diferentes elementos: 1) el rango de anomalías máximo-mínimo 2) el rango intercuartil 3) las anomalías del año 2018.
#rango de anomalías máximo-mínimo
g1.1 <- ggplot(data_norm)+
geom_crossbar(aes(x = mo, y = 0, ymin = min, ymax = mx),
fatten = 0, fill = "grey90", colour = "NA")
g1.1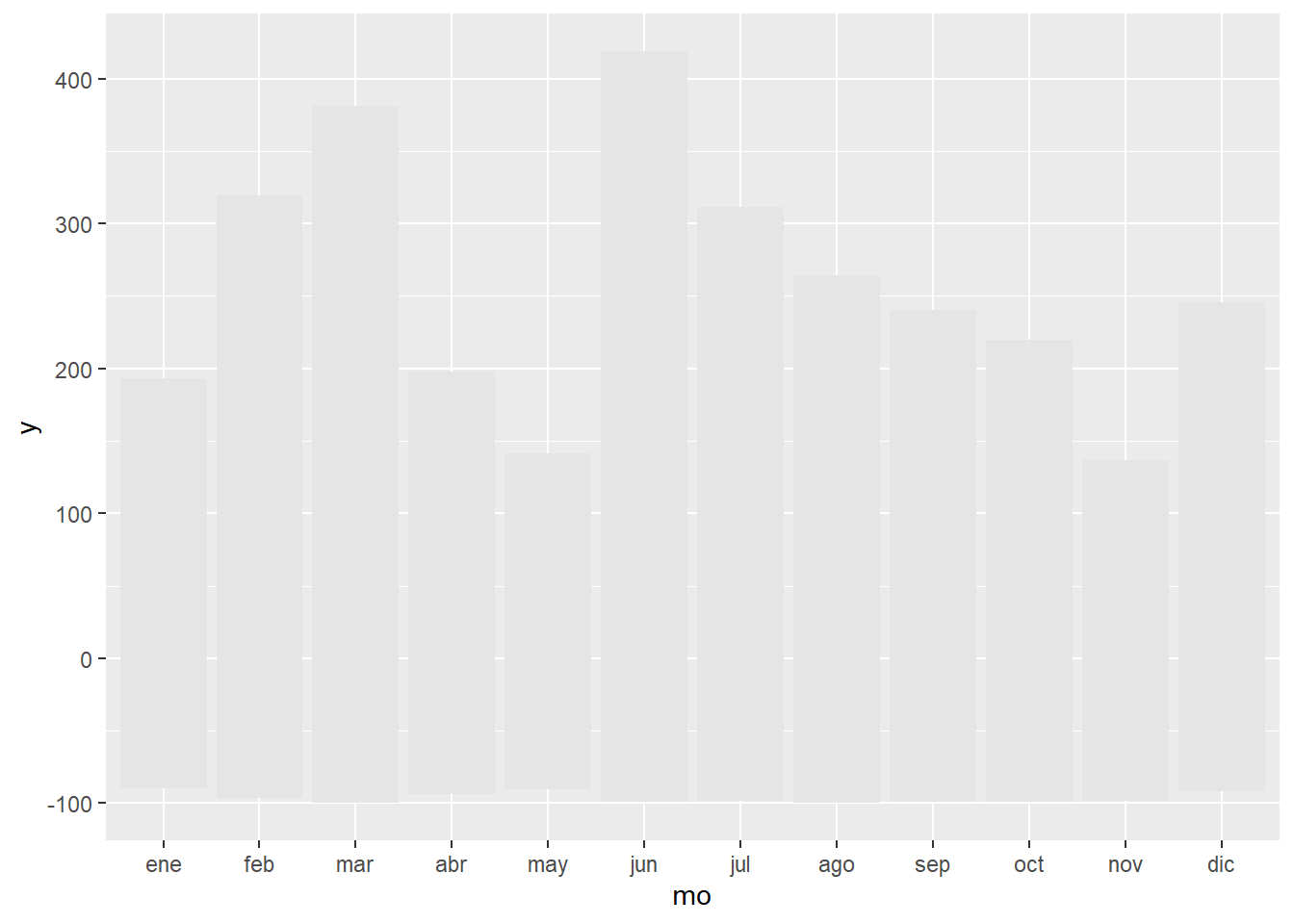
#añadinos el rango intercuartil
g1.2 <- g1.1 + geom_crossbar(aes(x = mo, y = 0, ymin = q25, ymax = q75),
fatten = 0, fill = "grey70")
g1.2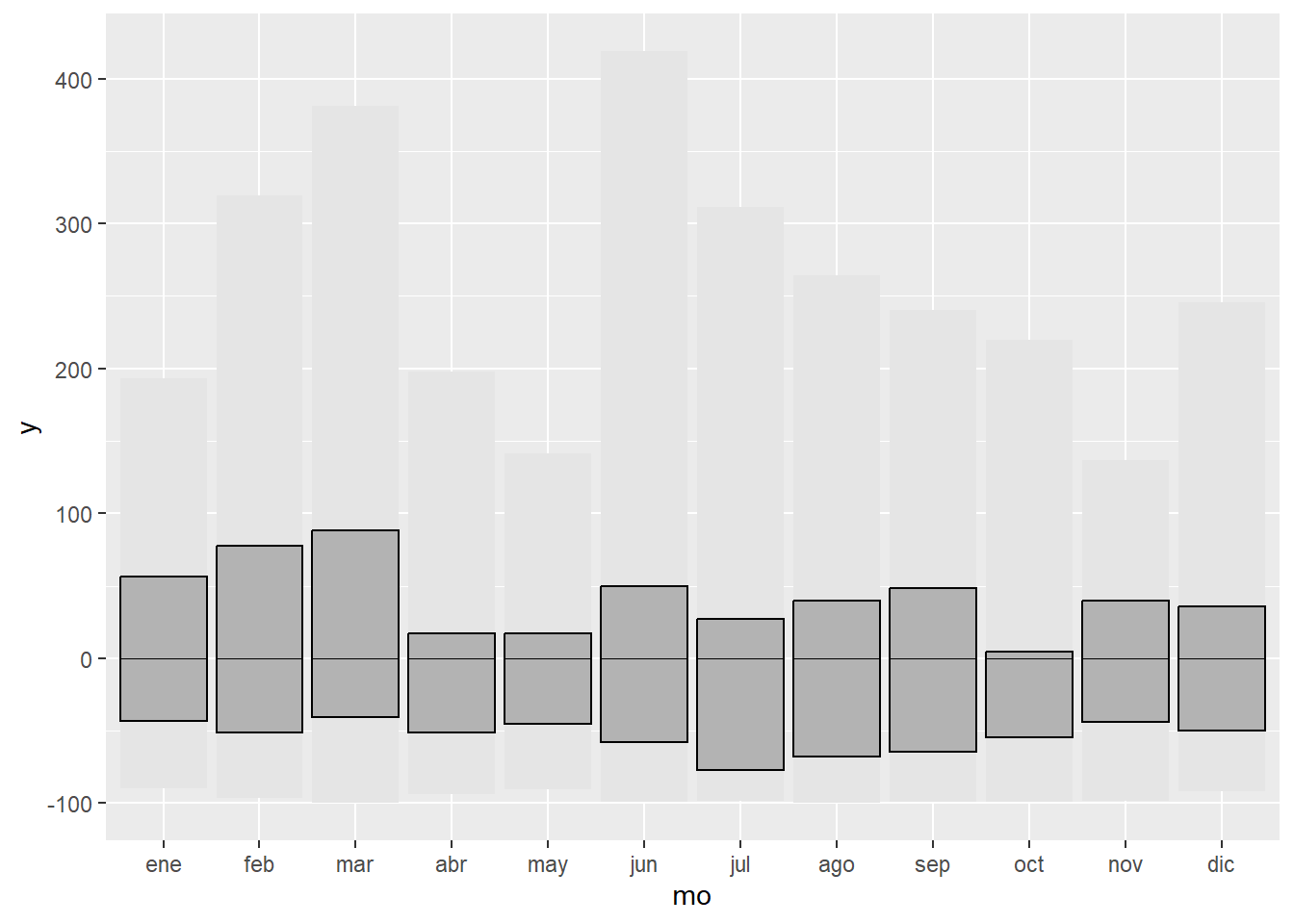
#añadimos las anomalías del año 2018
g1.3 <- g1.2 + geom_crossbar(data = filter(data, yr == 2018),
aes(x = mo, y = 0, ymin = 0, ymax = anom, fill = sign),
fatten = 0, width = 0.7, alpha = .7, colour = "NA",
show.legend = FALSE)
g1.3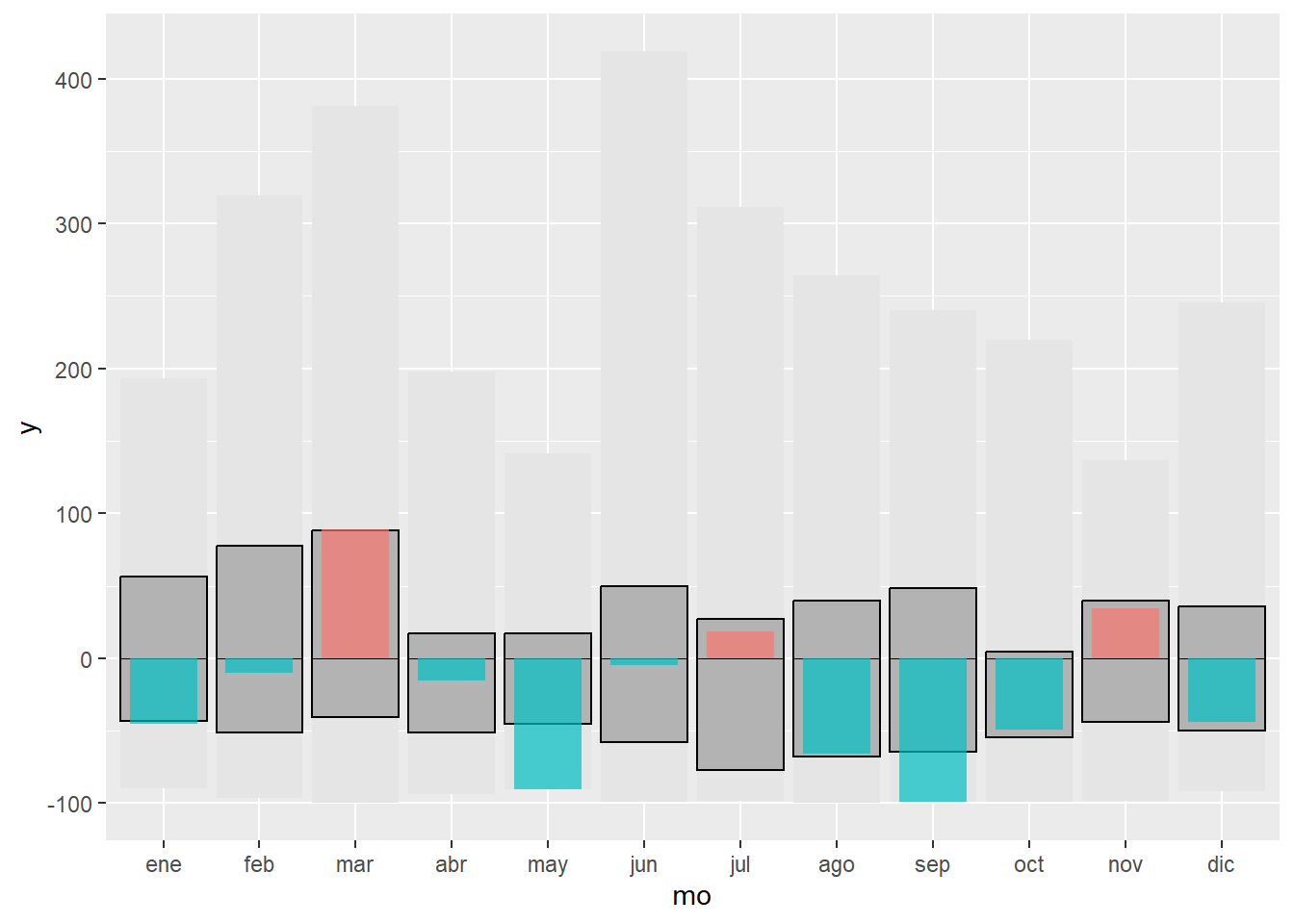
Finalmente añadimos unos últimos ajustes de estilo.
g1 <- g1.3 + geom_hline(yintercept = 0)+
scale_fill_manual(values=c("#99000d","#034e7b"))+
scale_y_continuous("Anomalía de precipitación (%)",
breaks = seq(-100, 500, 25),
expand = c(0, 5))+
labs(x = "",
title = "Anomalía de precipitación en Santiago de Compostela 2018",
caption="Dominic Royé (@dr_xeo) | Datos: eca.knmi.nl")+
theme_hc()
g1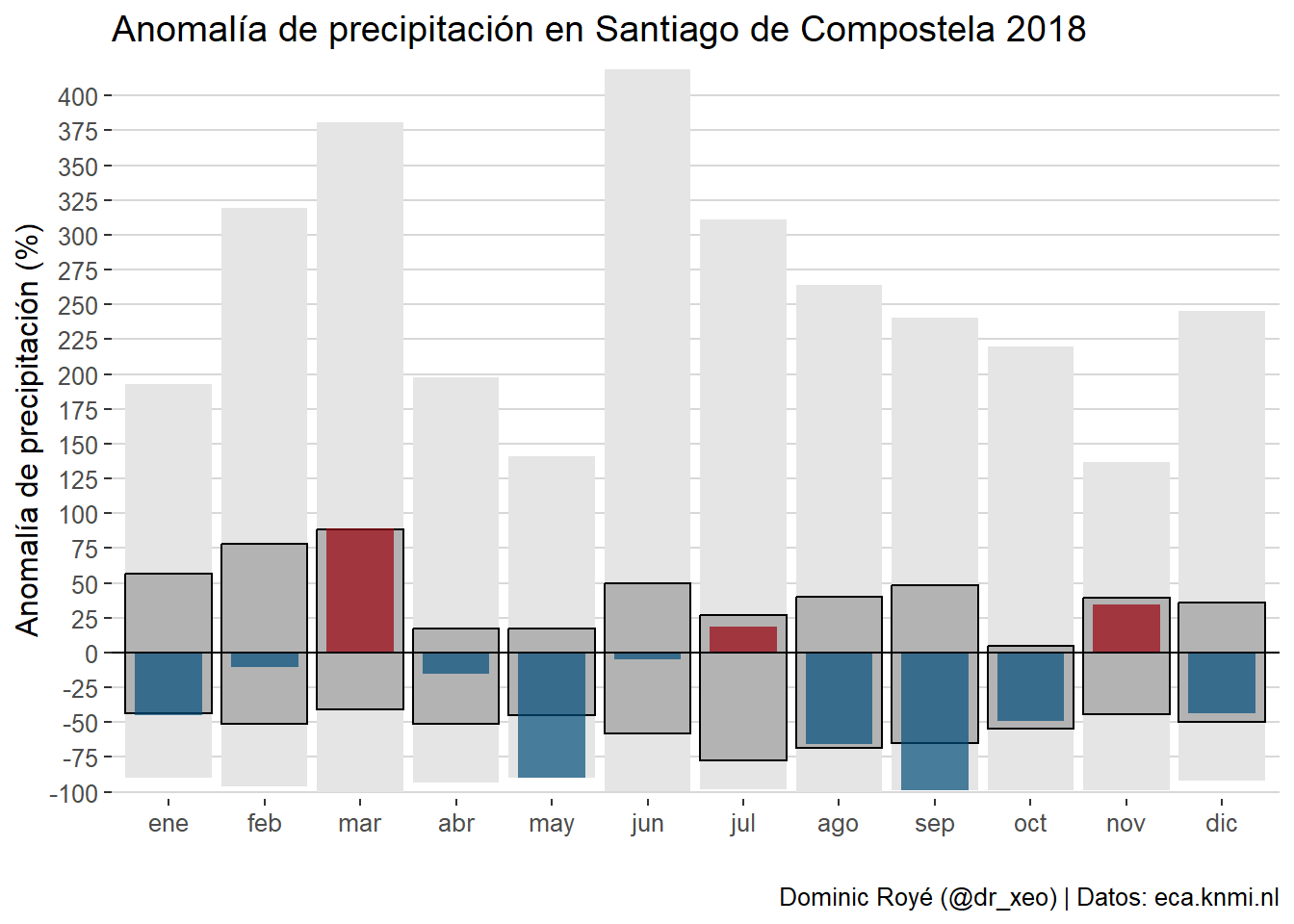
Parte 2
Todavía nos falta una leyenda. Primero la creamos para los valores normales.
#datos de la leyenda
legend <- filter(data_norm, mo == "ene")
legend_lab <- gather(legend, stat, y, mx:q75) %>%
mutate(stat = factor(stat, stat, c("máximo",
"mínimo",
"Cuantil 25%",
"Cuantil 75%")) %>%
as.character())## Warning: attributes are not identical across measure variables;
## they will be dropped#gráfico de la leyenda
g2 <- legend %>% ggplot()+
geom_crossbar(aes(x = mo, y = 0, ymin = min, ymax = mx),
fatten = 0, fill = "grey90", colour = "NA", width = 0.2) +
geom_crossbar(aes(x = mo, y = 0, ymin = q25, ymax = q75),
fatten = 0, fill = "grey70", width = 0.2) +
geom_text(data = legend_lab,
aes(x = mo, y = y+c(12,-8,-10,12), label = stat),
fontface = "bold", size = 2) +
annotate("text", x = 1.18, y = 40,
label = "Período 1950-2018", angle = 90, size = 3) +
theme_void() +
theme(plot.margin = unit(c(0, 0, 0, 0), "cm"))
g2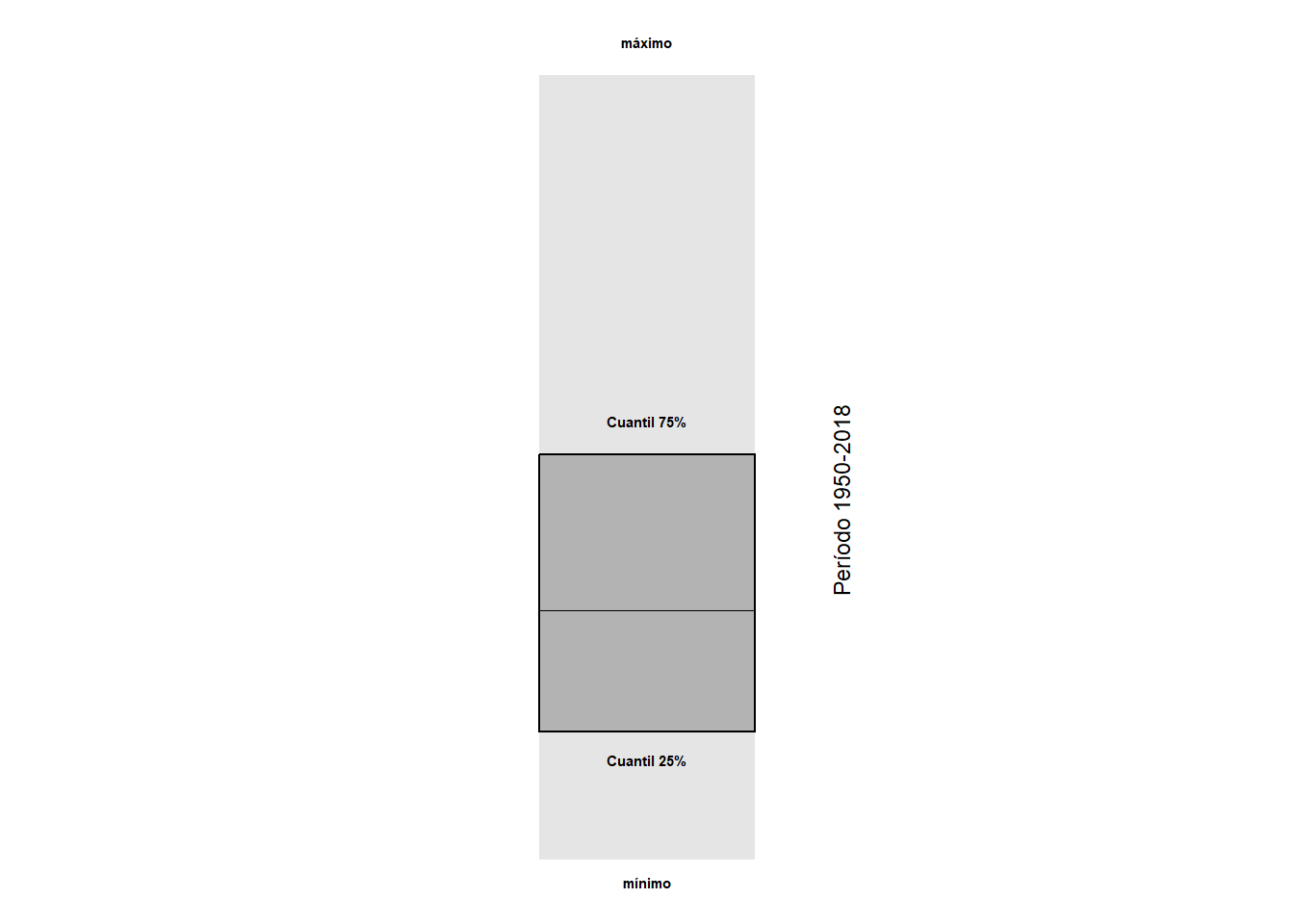
Segundo, creamos otra leyenda para las anomalías actuales.
legend2 <- filter(data, yr == 1950, mo %in% c("ene","feb")) %>%
ungroup() %>%
select(mo, anom, sign)
legend2[2,1] <- "ene"
legend_lab2 <- data.frame(mo = rep("ene", 3),
anom= c(110, 3, -70),
label = c("Anomalía positiva", "Promedio", "Anomalía negativa"))
g3 <- ggplot() +
geom_bar(data = legend2,
aes(x = mo, y = anom, fill = sign),
alpha = .6, colour = "NA", stat = "identity", show.legend = FALSE, width = 0.2) +
geom_segment(aes(x = .85, y = 0, xend = 1.15, yend = 0), linetype = "dashed") +
geom_text(data = legend_lab2, aes(x = mo, y = anom+c(10,5,-13), label = label), fontface = "bold", size = 2) +
annotate("text", x = 1.25, y = 20,
label ="Referencia 1971-2010", angle = 90, size = 3) +
scale_fill_manual(values = c("#99000d", "#034e7b")) +
theme_void() +
theme(plot.margin = unit(c(0, 0, 0, 0), "cm"))
g3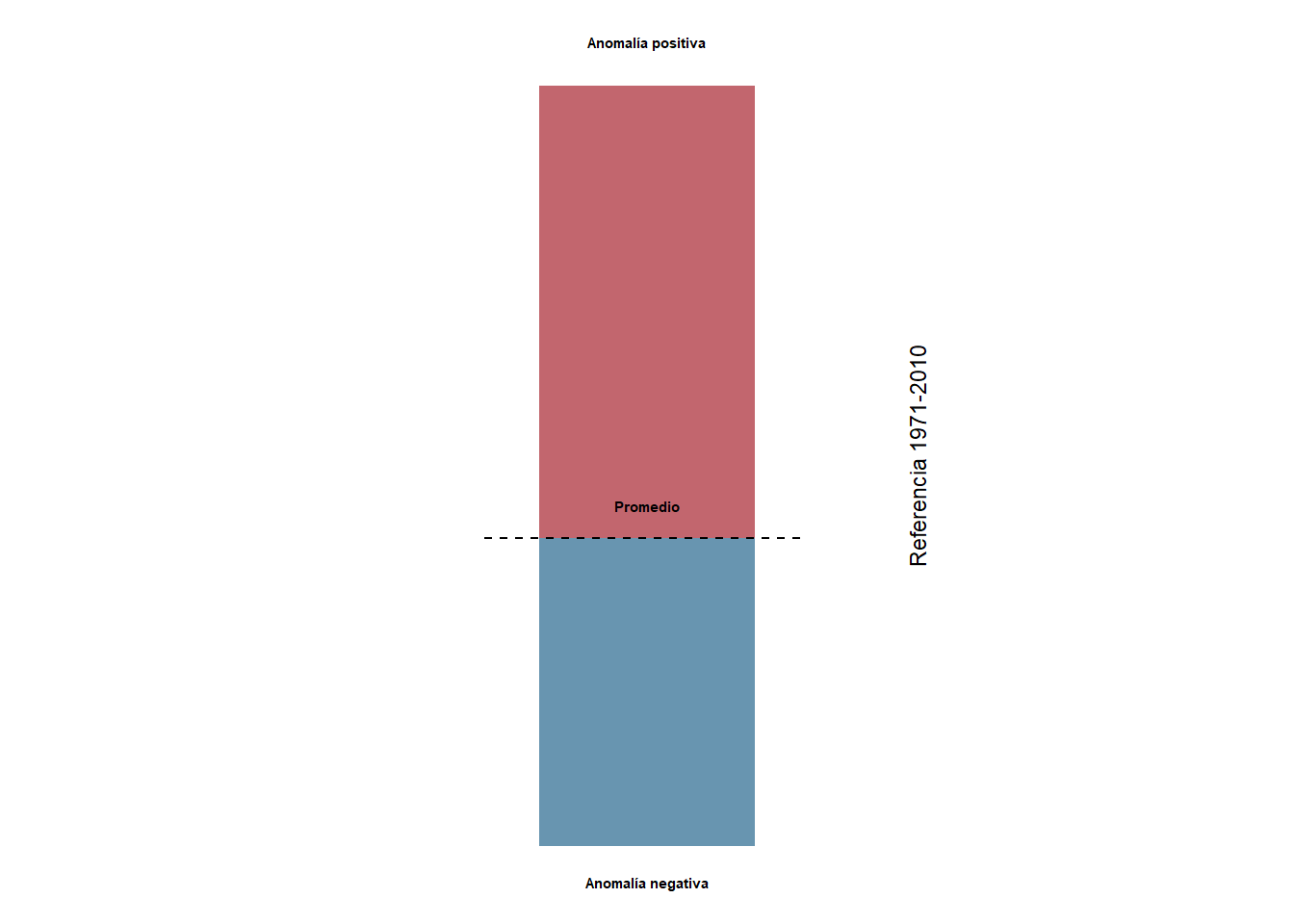
Parte 3
Para finalizar sólo debemos unir el gráfico y las leyendas con ayuda del paquete cowplot. La función princial de cowplot es plot_grid() que ayuda en combinar diferentes gráficos. No obstante, en este caso se hace necesario usar unas funciones más flexibles para crear formatos menos habituales. La función ggdraw() configura la capa básica del gráfico, y las funciones que están destinadas a operar en esta capa comienzan con draw_*.
p <- ggdraw() +
draw_plot(g1, x = 0, y = .3, width = 1, height = 0.6) +
draw_plot(g2, x = 0, y = .15, width = .2, height = .15) +
draw_plot(g3, x = 0.08, y = .15, width = .2, height = .15)
p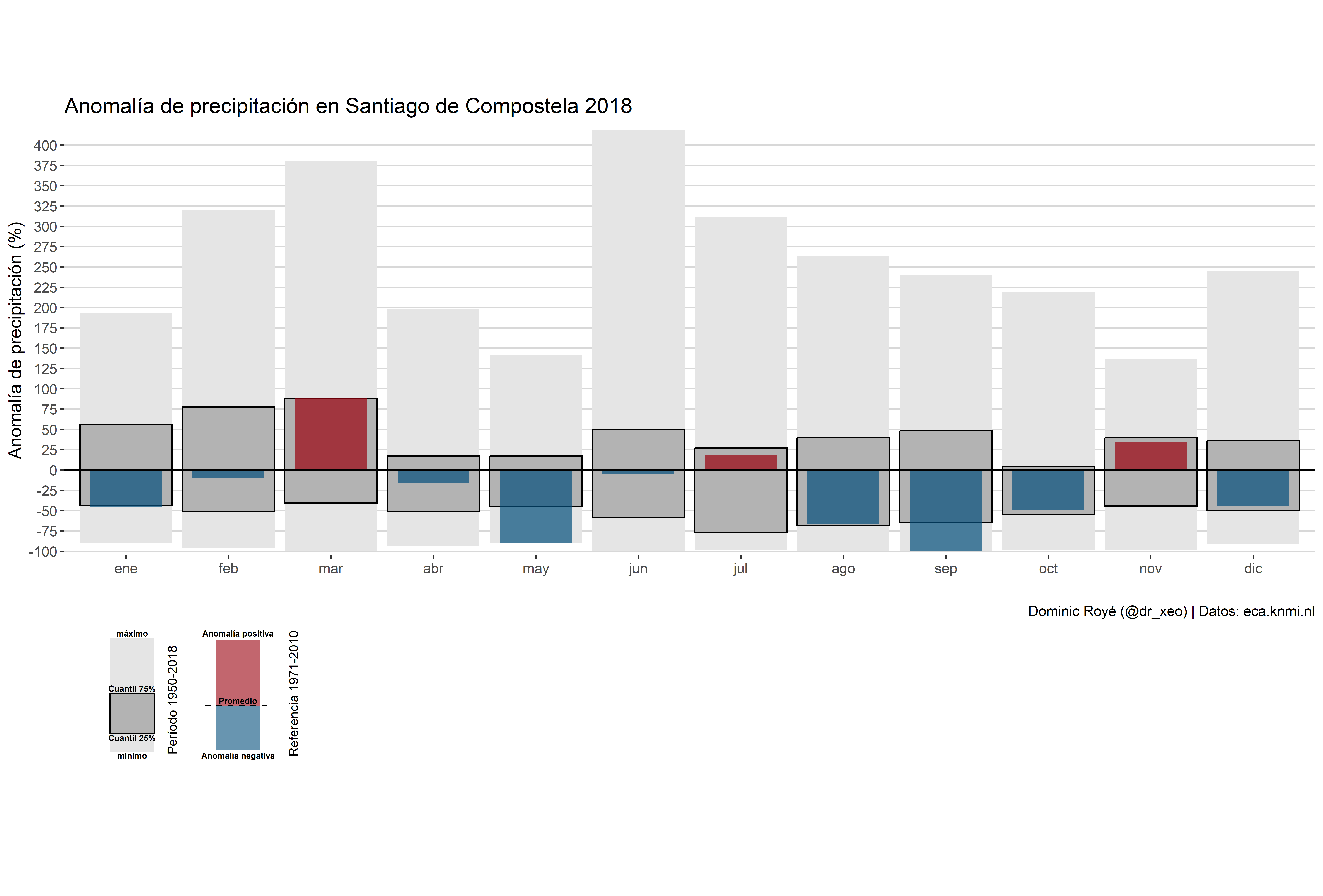
save_plot("pr_anomalia2016_scq.png", p, dpi = 300, base_width = 12.43, base_height = 8.42)Múltiples facetas
En este apartado haremos el mismo gráfico como en el anterior, pero para varios años.
Parte 1
Únicamente debemos filtrar por un conjunto de años, en este caso de 2016 a 2018, usando el operador %in%, además añadimos la función facet_grid() a ggplot, lo que nos permite plotear los gráficos según una variable. La formula usada para la función de facetas es similar al uso en modelos: variable_por_fila ~ variable_por_columna. Cuando no tenemos una variable en columna debemos usar el ..
#rango de anomalias maximo-minimo
g1.1 <- ggplot(data_norm)+
geom_crossbar(aes(x = mo, y = 0, ymin = min, ymax = mx),
fatten = 0, fill = "grey90", colour = "NA")
g1.1
#añadinos el rango intercuartil
g1.2 <- g1.1 + geom_crossbar(aes(x = mo, y = 0, ymin = q25, ymax = q75),
fatten = 0, fill = "grey70")
g1.2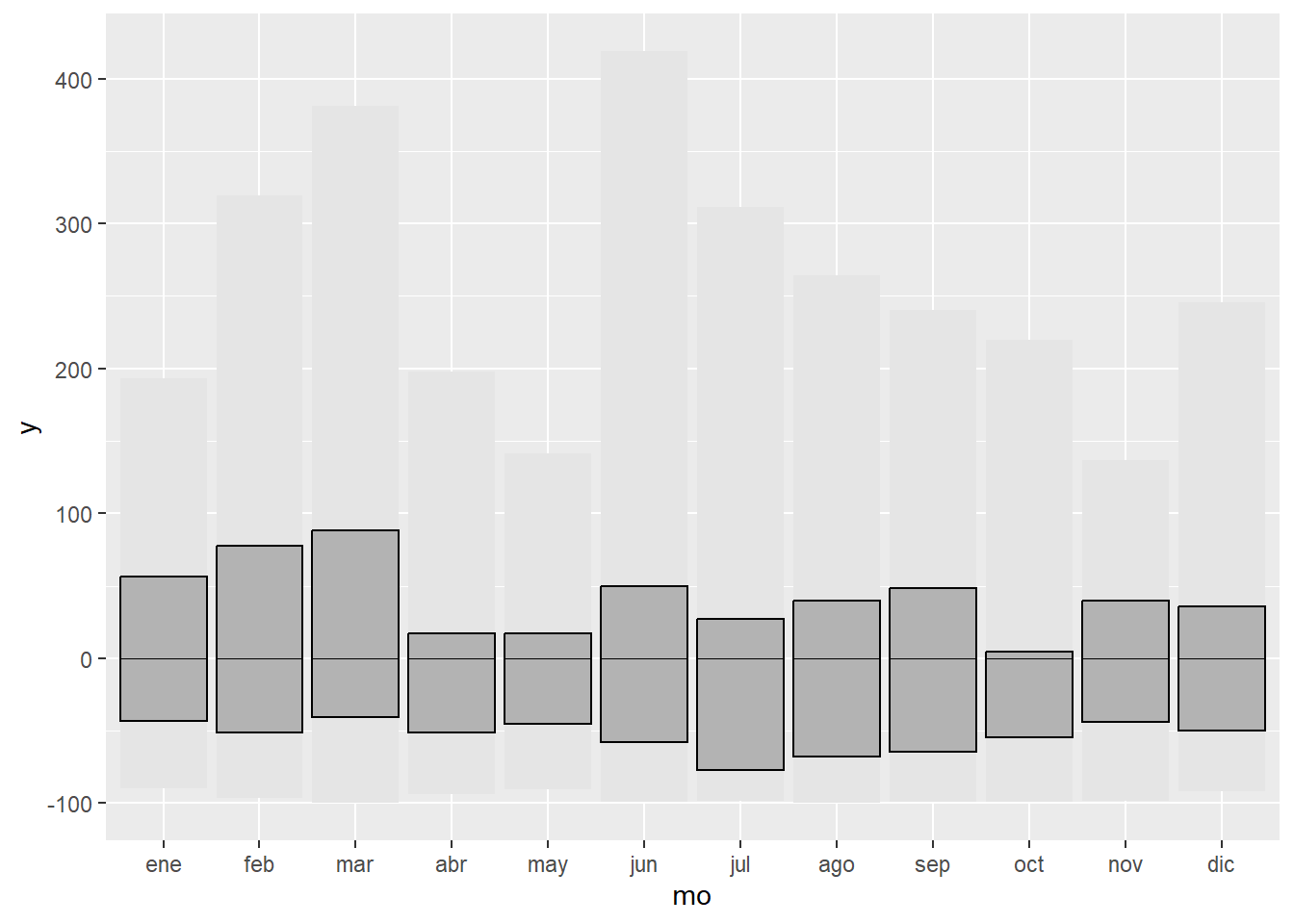
#añadimos las anomalías del año 2016-2018
g1.3 <- g1.2 + geom_crossbar(data = filter(data, yr %in% 2016:2018),
aes(x = mo, y = 0, ymin = 0, ymax = anom, fill = sign),
fatten = 0, width = 0.7, alpha = .7, colour = "NA",
show.legend = FALSE) +
facet_grid(yr ~ .)
g1.3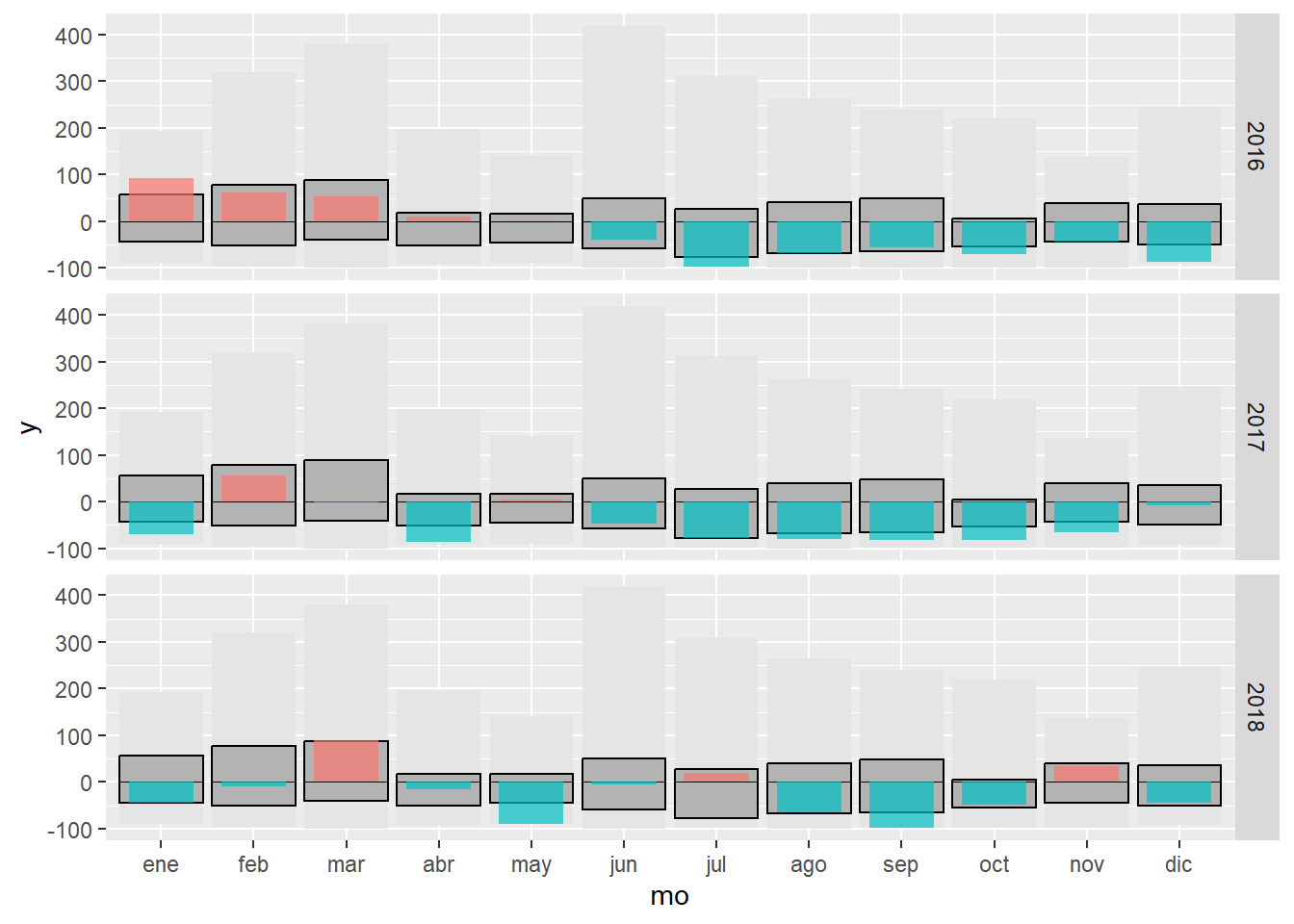
Finalmente, añadimos unos últimos ajustes de estilo.
g1 <- g1.3 + geom_hline(yintercept = 0)+
scale_fill_manual(values=c("#99000d","#034e7b"))+
scale_y_continuous("Anomalía de precipitación (%)",
breaks = seq(-100, 500, 50),
expand = c(0, 5))+
labs(x = "",
title = "Anomalía de precipitación en Santiago de Compostela",
caption="Dominic Royé (@dr_xeo) | Datos: eca.knmi.nl")+
theme_hc()
g1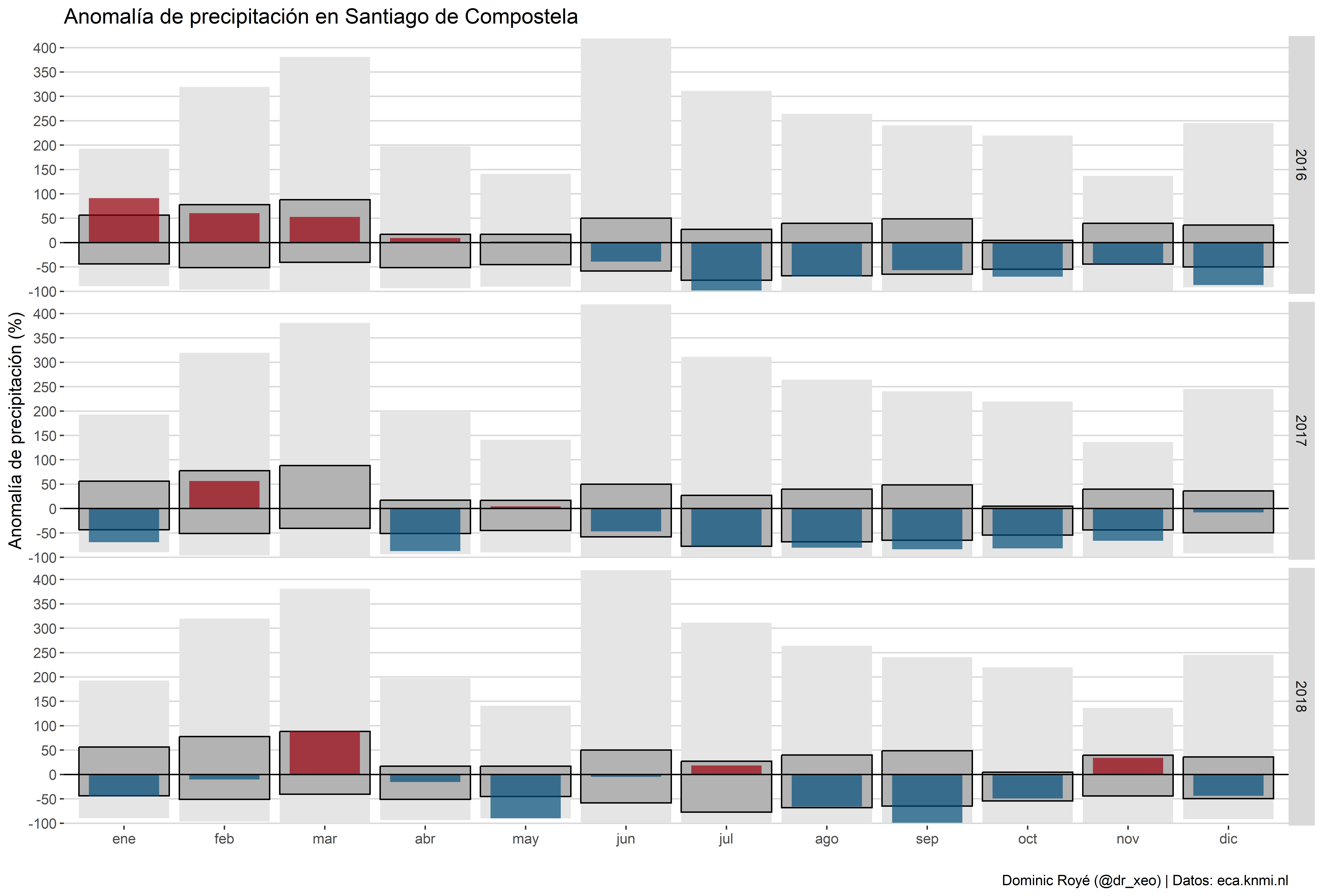
Usamos la misma leyenda universal creada para el gráfico anterior.
Parte 2
Para finalizar, sólo unimos el gráfico y las leyendas con ayuda del paquete cowplot. Lo único que debemos ajustar aquí son los argumentos en la función draw_plot() para colocar correctamente las diferentes partes.
p <- ggdraw() +
draw_plot(g1, x = 0, y = .18, width = 1, height = 0.8) +
draw_plot(g2, x = 0, y = .08, width = .2, height = .15) +
draw_plot(g3, x = 0.08, y = .08, width = .2, height = .15)
p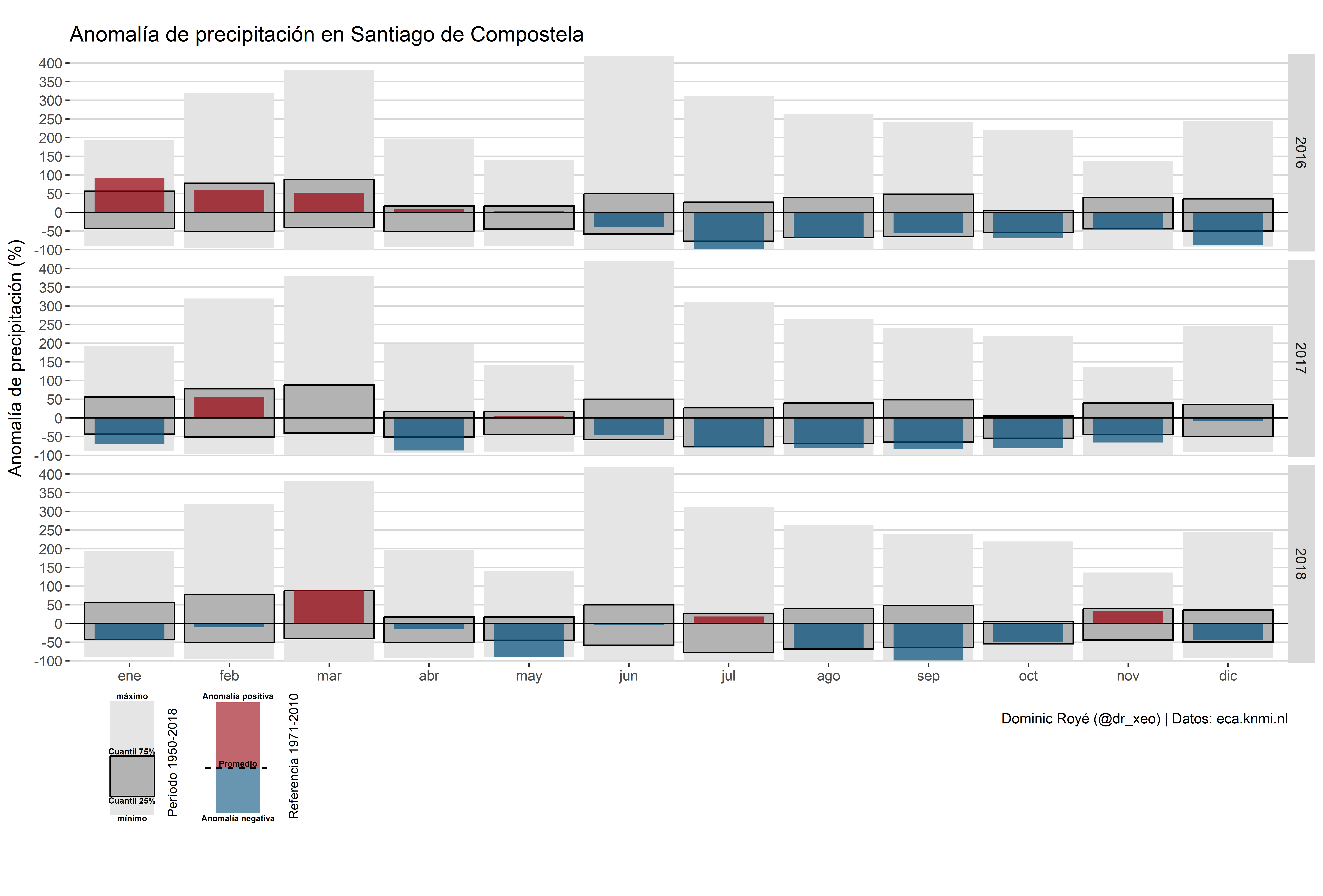
save_plot("pr_anomalia20162018_scq.png", p, dpi = 300, base_width = 12.43, base_height = 8.42)