Animación climática de la temperatura máxima
En el campo de la visualización de datos, la animación de datos espaciales en su dimensión temporal lleva a mostrar cambios y patrones fascinantes y muy visuales. A raíz de una de las últimas publicaciones que he realizado en los RRSS me pidieron que hiciera un post acerca de cómo lo creé. Pues bien, aquí vamos para empezar con datos de la España peninsular. Podéis encontrar más animaciones en la sección de gráficos de este mismo blog.
I couldn't resist to make another animation. Smoothed daily maximum temperature throughout the year in Europe. #rstats #ggplot2 #dataviz #climate pic.twitter.com/ZC9L0vh3vR
— Dr. Dominic Royé (@dr_xeo) May 9, 2020
Paquetes
En este post usaremos los siguientes paquetes:
| Paquete | Descripción |
|---|---|
| tidyverse | Conjunto de paquetes (visualización y manipulación de datos): ggplot2, dplyr, purrr,etc. |
| rnaturalearth | Mapas vectoriales del mundo ‘Natural Earth’ |
| lubridate | Fácil manipulación de fechas y tiempos |
| sf | Simple Feature: importar, exportar y manipular datos vectoriales |
| raster | Importar, exportar y manipular raster |
| ggthemes | Estilos para ggplot2 |
| gifski | Crear gifs |
| showtext | Usar fuentes más fácilmente en gráficos R |
| sysfonts | Cargar fuentes del sistema y fuentes de Google |
# instalamos los paquetes si hace falta
if(!require("tidyverse")) install.packages("tidyverse")
if(!require("rnaturalearth")) install.packages("rnaturalearth")
if(!require("lubridate")) install.packages("lubridate")
if(!require("sf")) install.packages("sf")
if(!require("ggthemes")) install.packages("ggthemes")
if(!require("gifski")) install.packages("gifski")
if(!require("raster")) install.packages("raster")
if(!require("sysfonts")) install.packages("sysfonts")
if(!require("showtext")) install.packages("showtext")
# paquetes
library(raster)
library(tidyverse)
library(lubridate)
library(ggthemes)
library(sf)
library(rnaturalearth)
library(extrafont)
library(showtext)
library(RColorBrewer)
library(gifski)Para aquellos con menos experiencia con tidyverse, recomiendo la breve introducción en este blog post.
Preparación
Datos
Descargamos los datos STEAD de la temperatura máxima (tmax_pen.nc) en formato netCDF desde el repositario del CSIC aquí (el tamaño de los datos es de 2 GB). Se trata de un conjunto de datos con una resolución espacial de 5 km y comprenden las temperaturas máximas diarias desde 1901 a 2014. En la climatología y la meteorología, un formato de uso muy extendido es el de las bases de datos netCDF, que permiten obtener una estructura multidimensional e intercambiar los datos de forma independiente al sistema operativo empleado. Se trata de un formato espacio-temporal con una cuadrícula regular o irregular. La estructura multidimensional en forma de matriz (array) permite usar no sólo datos espacio-temporales sino también multivariables. En nuestros datos tendremos un cubo de tres dimensiones: longitud, latitud y tiempo de la temperatura máxima.

Royé 2015. Sémata: Ciencias Sociais e Humanidades 27:11-37
Importar los datos
El formato netCDF con extensión .nc lo podemos importar vía dos paquetes principales: 1) ncdf4 y 2) raster. Aunque el paquete raster realmente lo que hace es usar el primer paquete para importar los datos. En este post usaremos el paquete raster dado que es algo más fácil, con algunas funciones muy útiles y más universales para todo tipo de formato raster. Las funciones principales de importación son: raster(), stack() y brick(). La primera función sólo permite importar una única capa, en cambio, las últimas dos funciones se emplean para datos multidimensionales. En nuestro caso sólo tenemos una variable, por tanto no sería necesario hacer uso del argumento varname.
# importamos los datos ncdf
tmx <- brick("tmax_pen.nc", varname = "tx")## Loading required namespace: ncdf4tmx # metadatos## class : RasterBrick
## dimensions : 190, 230, 43700, 41638 (nrow, ncol, ncell, nlayers)
## resolution : 0.0585, 0.045 (x, y)
## extent : -9.701833, 3.753167, 35.64247, 44.19247 (xmin, xmax, ymin, ymax)
## crs : +proj=longlat +datum=WGS84 +no_defs
## source : tmax_pen.nc
## names : X1, X2, X3, X4, X5, X6, X7, X8, X9, X10, X11, X12, X13, X14, X15, ...
## Time (days since 1901-01-01): 1, 41638 (min, max)
## varname : txEn la estructura del objeto RasterBrick vemos todos los metadatos necesarios: desde la resolución, las dimensiones o el tipo de proyección, hasta el nombre de la variable. Además nos indica que únicamente apunta a los datos (source) y no los ha importado a la memoria RAM lo que facilita el trabajo con grandes conjuntos de datos.
Para acceder a cualquier capa hacemos uso de [[ ]] con el índice correspondiente. Así podemos plotear fácilmente cualquier día de los 41.638 días de los que disponemos.
# mapear cualquier día
plot(tmx[[200]], col = rev(heat.colors(7)))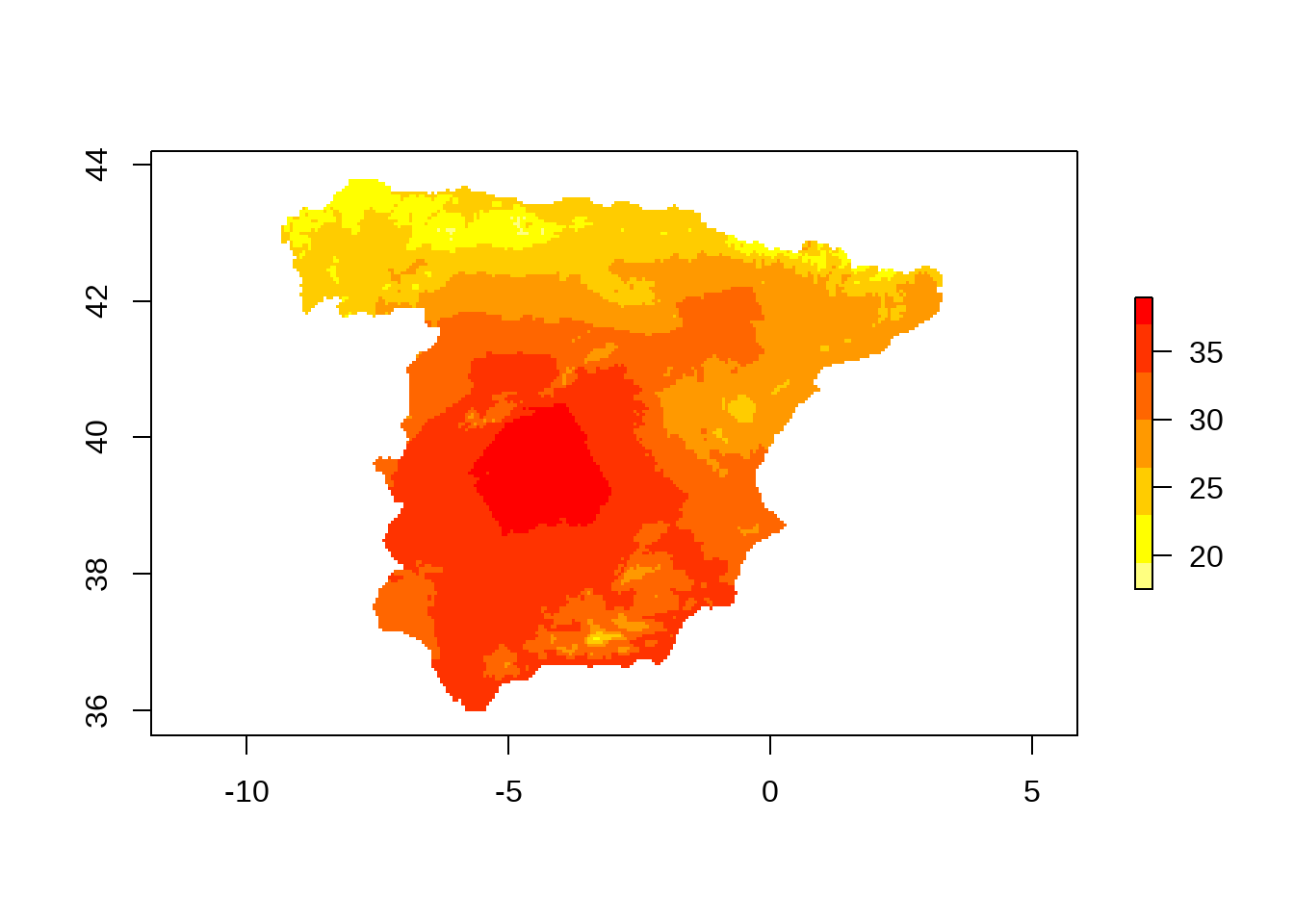
Calcular el promedio de la temperatura
En este paso el objetivo es calcular el promedio de la temperatura para cada día del año. Por eso, lo primero que hacemos es crear un vector, indicando el día del año para toda la serie temporal. En el paquete raster disponemos de la función stackApply() que permite aplicar una función sobre grupos de capas, o mejor dicho, índices. Dado que nuestro conjunto de datos es grande, incluimos esta función en las funciones de paralelización.
Empezamos con las funciones beginClusterr() y endCluster() que inician y finalizan la paralelización. En la primera debemos indicar el número de núcleos que queremos usar. En este caso uso 4 de 7 posibles núcleos, no obstante, se debe cambiar el número según las características de cada CPU, siendo la norma n-1. Entonces, la función clusterR permite ejecutar funciones en paralelo con múltiples núcleos. El primer argumento corresponde al objeto raster, el segundo a la función empleada, y en forma de lista pasamos los argumentos de la función stackApply(), los índices que crean los grupos y la función usada para cada uno de los grupos. Si añadimos el argumento progress = 'text' se muestra una barra de progreso del cálculo.
# convertimos las fechas entre 1901 y 2014 a días del año
time_days <- yday(seq(as_date("1901-01-01"), as_date("2014-12-31"), "day"))
# calculamos el promedio
beginCluster(4)
tmx_mean <- clusterR(tmx, stackApply, args = list(indices = time_days, fun = mean))
endCluster()Suavizar la variabilidad de las temperaturas
Antes de pasar a suavizar las series temporales de nuestro RasterBrick, un ejemplo del por qué lo hacemos. Extraemos un píxel de nuestro conjunto de datos en las coordenadas -1º de longitud y 40º de latitud usando la función extract(). Dado que la función con el mismo nombre aparece en varios paquetes, debemos cambiar a la forma nombre_paquete::nombre_función. El resultado es una matriz con una fila correspondiente al píxel y 366 columnas de los días del año. El siguiente paso es la creación de un data.frame con una fecha dummy y la temperatura máxima extraída.
# extraemos un píxel
point_ts <- raster::extract(tmx_mean, matrix(c(-1, 40), nrow = 1))
dim(point_ts) # dimensiones ## [1] 1 366# creamos un data.frame
df <- data.frame(date = seq(as_date("2000-01-01"), as_date("2000-12-31"), "day"),
tmx = point_ts[1,])
# visualizamos la temperatura máxima
ggplot(df,
aes(date, tmx)) +
geom_line() +
scale_x_date(date_breaks = "month", date_labels = "%b") +
scale_y_continuous(breaks = seq(5, 28, 2)) +
labs(y = "Temperatura máxima", x = "", colour = "") +
theme_minimal()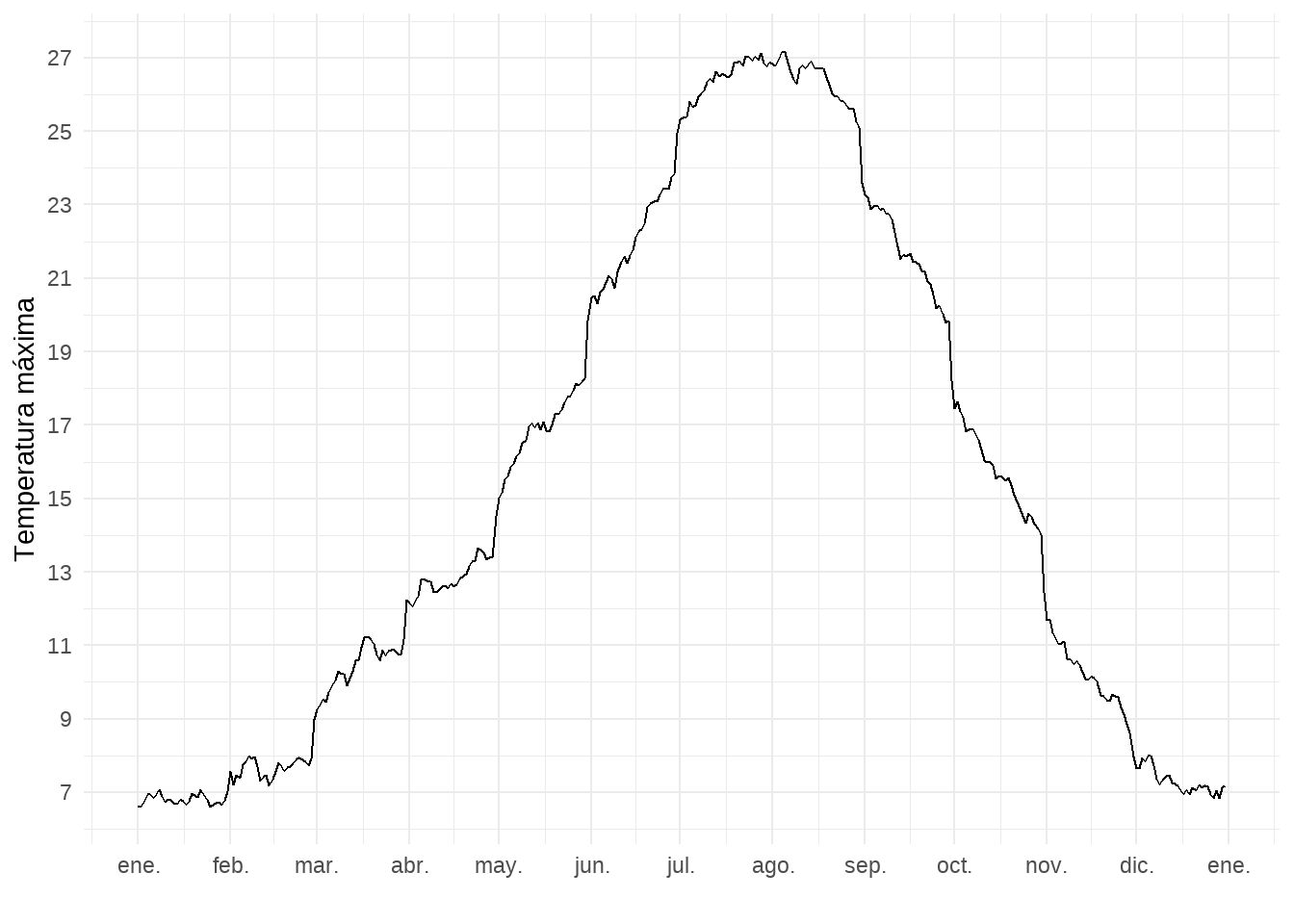
El gráfico muestra claramente la todavía existente variabilidad, lo que haría fluctuar bastante una animación. Por eso, creamos una función de suavizado basado en un ajuste de regresión polinomial local (LOESS), más detalles los encontráis en la ayuda de la función loess(). El argumento más importante es span que determina el grado de suavizado de la función, cuanto más pequeño el valor menos suave será la curva. El mejor resultado me ha dado un valor del 0,5.
daily_smooth <- function(x, span = 0.5){
if(all(is.na(x))){
return(x)
} else {
df <- data.frame(yd = 1:366, ta = x)
m <- loess(ta ~ yd, span = span, data = df)
est <- predict(m, 1:366)
return(est)
}
}Aplicamos nuestra nueva función de suavizado a la serie temporal extraída y hacemos algunos cambios para poder visualizar la diferencia entre los datos originales y suavizados.
# suavizamos la temperatura
df <- mutate(df, tmx_smoothed = daily_smooth(tmx)) %>%
pivot_longer(2:3, names_to = "var", values_to = "temp")
# visualizamos la diferencia
ggplot(df,
aes(date, temp,
colour = var)) +
geom_line() +
scale_x_date(date_breaks = "month", date_labels = "%b") +
scale_y_continuous(breaks = seq(5, 28, 2)) +
scale_colour_manual(values = c("#f4a582", "#b2182b")) +
labs(y = "Temperatura máxima", x = "", colour = "") +
theme_minimal()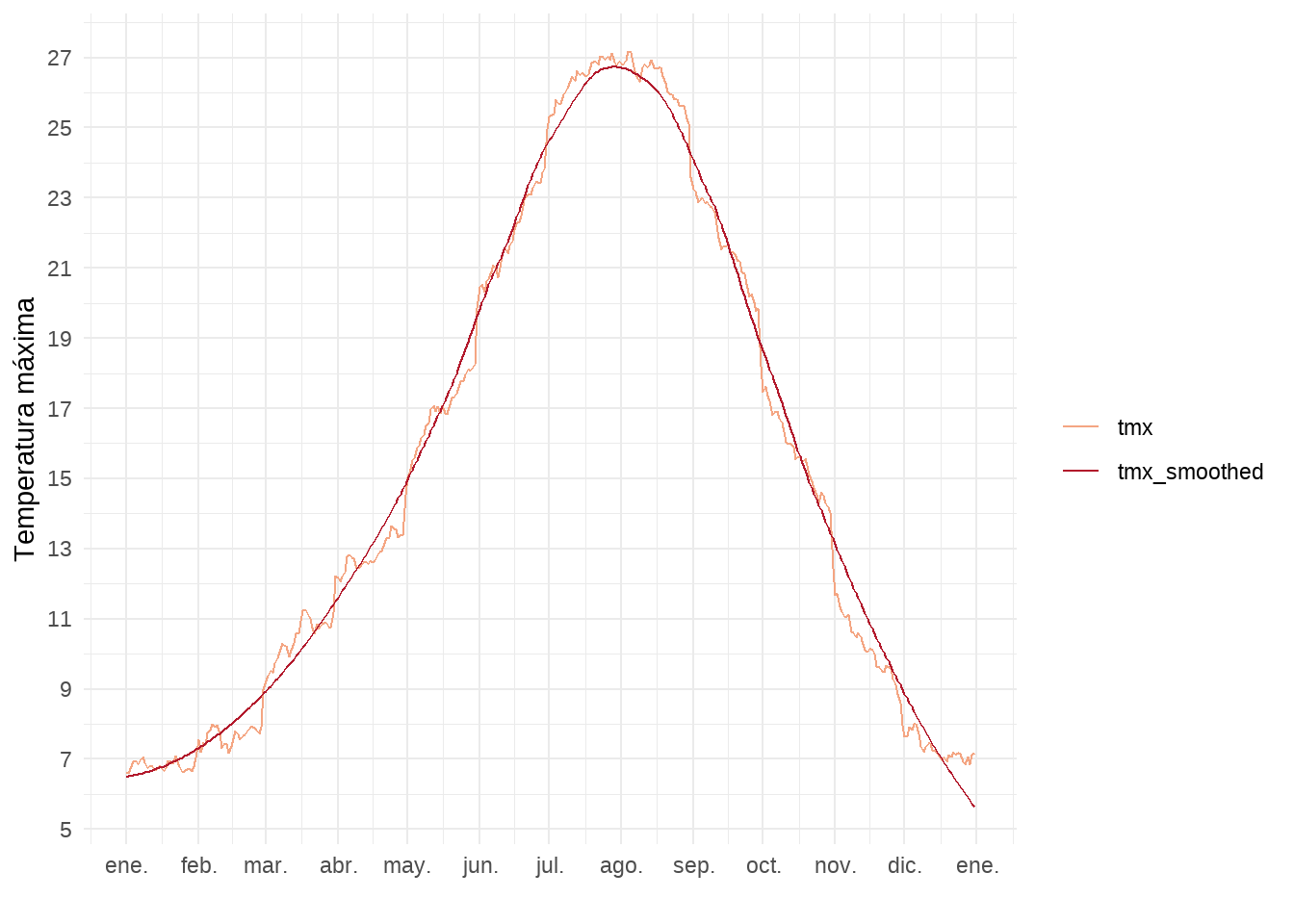
Como vemos en el gráfico la curva suavizada sigue muy bien la curva original. En el siguiente paso empleamos nuestra función sobre el RasterBrick usando la función calc(). La función devuelve tantas capas como las que devuelve la función empleada a cada de las series temporales.
# suavizar el RasterBrick
tmx_smooth <- calc(tmx_mean, fun = daily_smooth)Visualización
Preparación
Para visualizar las temperaturas máximas durante todo el año, primero, convertimos el RasterBrick a un data.frame, incluyendo longitud y latitud, pero eliminando todas las series temporales sin valores (NA).
# convertir a data.frame
tmx_mat <- as.data.frame(tmx_smooth, xy = TRUE, na.rm = TRUE)
# renombrar las columnas
tmx_mat <- set_names(tmx_mat, c("lon", "lat", str_c("D", 1:366)))
str(tmx_mat[, 1:10])## 'data.frame': 20676 obs. of 10 variables:
## $ lon: num -8.03 -7.98 -7.92 -7.86 -7.8 ...
## $ lat: num 43.8 43.8 43.8 43.8 43.8 ...
## $ D1 : num 10.5 10.3 10 10.9 11.5 ...
## $ D2 : num 10.5 10.3 10.1 10.9 11.5 ...
## $ D3 : num 10.5 10.3 10.1 10.9 11.5 ...
## $ D4 : num 10.6 10.4 10.1 10.9 11.5 ...
## $ D5 : num 10.6 10.4 10.1 11 11.6 ...
## $ D6 : num 10.6 10.4 10.1 11 11.6 ...
## $ D7 : num 10.6 10.4 10.2 11 11.6 ...
## $ D8 : num 10.6 10.4 10.2 11 11.6 ...Segundo, importamos los límites administrativos con la función ne_countries() del paquete rnaturalearth limitando la extensión a la región de la Península Ibérica, el sur de Francia y el norte de África.
# importamos los límites globales
map <- ne_countries(scale = 10, returnclass = "sf") %>% st_cast("MULTILINESTRING")
# limitamos la extensión
map <- st_crop(map, xmin = -10, xmax = 5, ymin = 35, ymax = 44) ## Warning: attribute variables are assumed to be spatially constant throughout all
## geometries# mapa de los límites
plot(map)## Warning: plotting the first 9 out of 94 attributes; use max.plot = 94 to plot
## all
Tercero, creamos un vector con etiquetas del día del año para incluirlas en la animación. Además, definimos los cortes de la temperatura máxima, adaptados a la distribución de nuestros datos, para obtener una categorización con un total de 20 clases.
Cuarto, aplicamos la función cut() con los cortes a todas las columnas con las temperaturas de cada día del año.
# etiquetas de los días del año
lab <- as_date(0:365, "2000-01-01") %>% format("%d %B")
# cortes para la temperatura
ct <- c(-5, 0, 4, 6, 8, 10, 12, 14, 16, 18, 20, 22, 24, 26, 28, 30, 32, 34, 40, 45)
# datos categorizados con los cortes fijados
tmx_mat_cat <- mutate_at(tmx_mat, 3:368, cut, breaks = ct)Quinto, descargamos la fuente Montserrat y definimos los colores correspondientes a las clases creadas.
# descarga de la fuente
font_add_google("Montserrat", "Montserrat")
# uso de showtext con DPI 300
showtext_opts(dpi = 300)
showtext_auto()
# definimos una rampa de colores
col_spec <- colorRampPalette(rev(brewer.pal(11, "Spectral")))Mapa estático
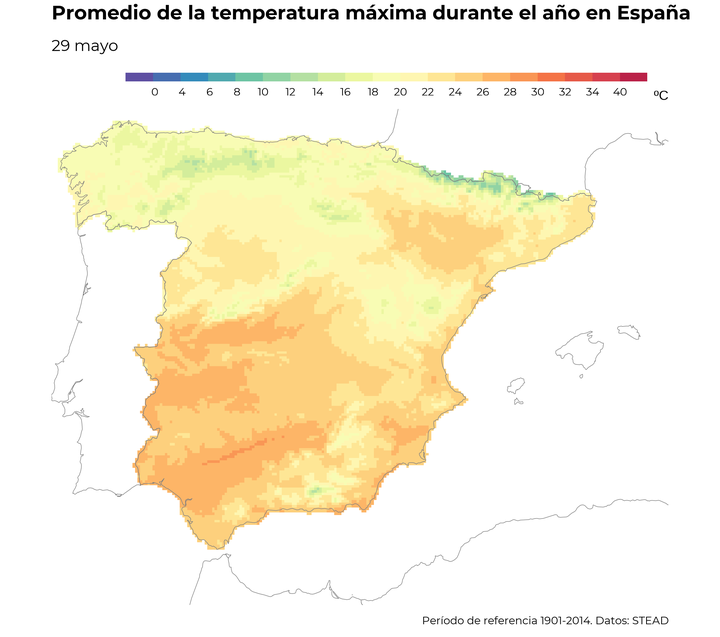
En esta primera visualización hacemos un mapa del 29 de mayo (día 150). No voy a explicar todos los detalles de la construcción con ggplot2, no obstante, es importante destacar que hago uso de la función aes_string() en lugar de aes() para poder usar los nombres de las columnas en formato de carácter. Con la función geom_raster() añadimos los datos en rejilla de temperatura como primera capa del gráfico y con geom_sf() los límites de clase sf. Por último, la función guide_colorsteps() permite crear una bonita leyenda basada en las clases creadas por la función cut().
ggplot(tmx_mat_cat) +
geom_raster(aes_string("lon", "lat", fill = "D150")) +
geom_sf(data = map,
colour = "grey50", size = 0.2) +
coord_sf(expand = FALSE) +
scale_fill_manual(values = col_spec(20), drop = FALSE) +
guides(fill = guide_colorsteps(barwidth = 30,
barheight = 0.5,
title.position = "right",
title.vjust = .1)) +
theme_void() +
theme(legend.position = "top",
legend.justification = 1,
plot.caption = element_text(family = "Montserrat",
margin = margin(b = 5, t = 10, unit = "pt")),
plot.title = element_text(family = "Montserrat",
size = 16, face = "bold",
margin = margin(b = 2, t = 5, unit = "pt")),
legend.text = element_text(family = "Montserrat"),
plot.subtitle = element_text(family = "Montserrat",
size = 13,
margin = margin(b = 10, t = 5, unit = "pt"))) +
labs(title = "Promedio de la temperatura máxima durante el año en España",
subtitle = lab[150],
caption = "Período de referencia 1901-2014. Datos: STEAD",
fill = "ºC")
Animación de todo el año
La animación final consiste en crear un gif a partir de todas las imágenes de los 366 días, en principio, se podría usar el paquete gganimate, pero en mi experiencia es más lento, dado que requiere un data.frame en formato largo. En este ejemplo una tabla larga tendría más de siete millones de filas, por eso, lo que hacemos es usar un bucle sobre las columnas y unir todas las imágenes creadas con el paquete gifski que también usa gganimate para la reproducción en formato gif.
Antes del bucle creamos un vector con los pasos temporales o nombres de las columnas y otro vector con el nombre de las imágenes, incluida el nombre de la carpeta. Con el objetivo de obtener una lista de imágenes ordenadas por su número debemos mantener tres cifras rellenando las posiciones a la izquierda con ceros.
time_step <- str_c("D", 1:366)
files <- str_c("./ta_anima/D", str_pad(1:366, 3, "left", "0"), ".png")Por último, incluimos la construcción anterior del gráfico en un bucle for.
for(i in 1:366){
ggplot(tmx_mat_cat) +
geom_raster(aes_string("lon", "lat", fill = time_step[i])) +
geom_sf(data = map,
colour = "grey50", size = 0.2) +
coord_sf(expand = FALSE) +
scale_fill_manual(values = col_spec(20), drop = FALSE) +
guides(fill = guide_colorsteps(barwidth = 30,
barheight = 0.5,
title.position = "right",
title.vjust = .1)) +
theme_void() +
theme(legend.position = "top",
legend.justification = 1,
plot.caption = element_text(family = "Montserrat",
margin = margin(b = 5, t = 10, unit = "pt")),
plot.title = element_text(family = "Montserrat",
size = 16, face = "bold",
margin = margin(b = 2, t = 5, unit = "pt")),
legend.text = element_text(family = "Montserrat"),
plot.subtitle = element_text(family = "Montserrat",
size = 13,
margin = margin(b = 10, t = 5, unit = "pt"))) +
labs(title = "Promedio de la temperatura máxima durante el año en España",
subtitle = lab[i],
caption = "Período de referencia 1901-2014. Datos: STEAD",
fill = "ºC")
ggsave(files[i], width = 8.28, height = 7.33, type = "cairo")
}Después de haber creado imágenes para cada día del año, únicamente nos queda por crear el gif.
gifski(files, "tmx_spain.gif", width = 800, height = 700, loop = FALSE, delay = 0.05)