Visualizar anomalías climáticas
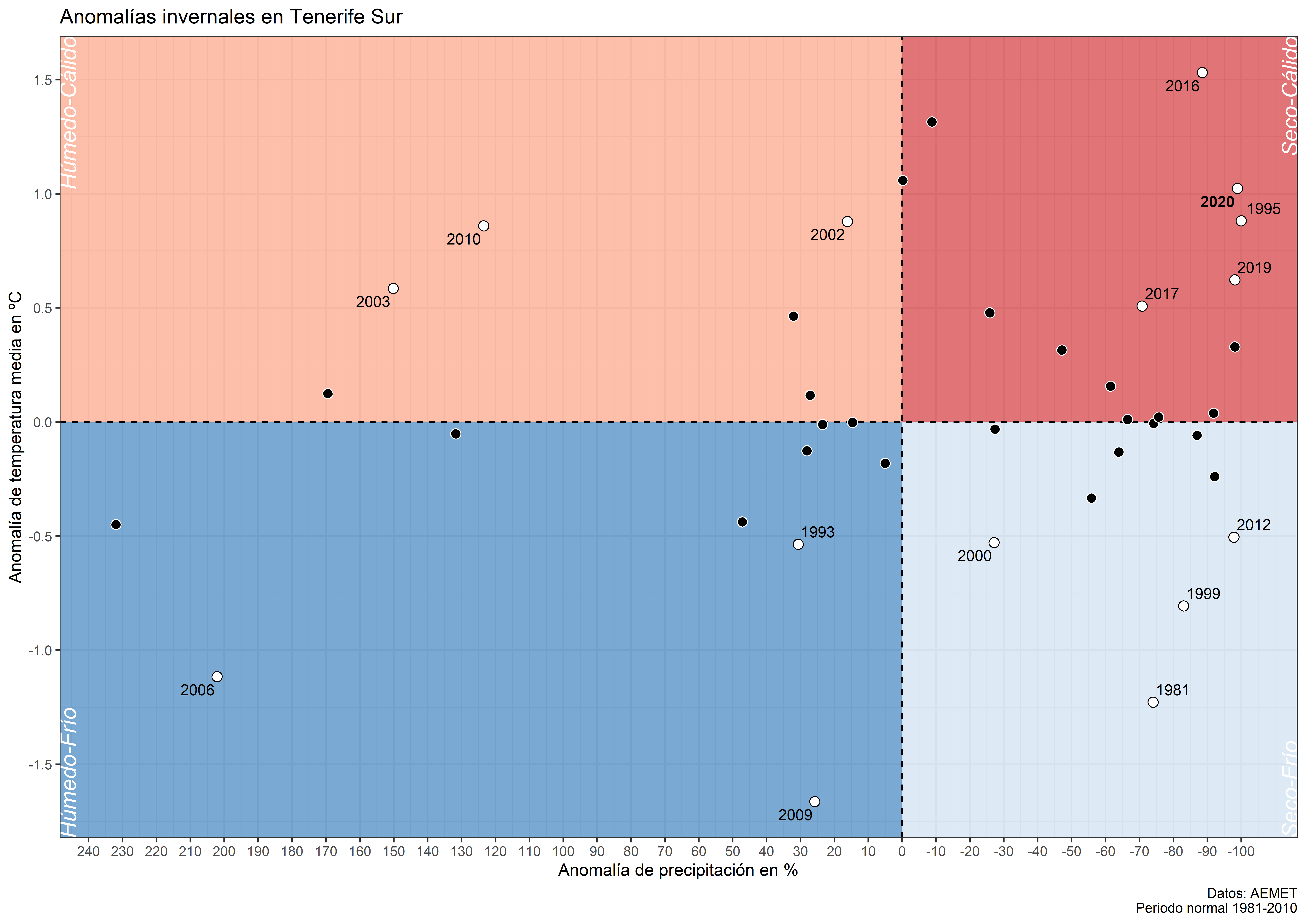
Cuando visualizamos anomalías de precipitación y temperatura, simplemente usamos series temporales en un gráfico de barras indicando con color rojo y azul valores negativos y positivos. No obstante, para tener una imagen global necesitamos ambas anomalías en un único gráfico. Así podríamos responder directamente a la pregunta de si una estación del año o un mes en concreto fue seco-cálido o húmedo-frío, e incluso comparar estas anomalías en el contexto de años anteriores.
Paquetes
En este post usaremos los siguientes paquetes:
| Paquete | Descripción |
|---|---|
| tidyverse | Conjunto de paquetes (visualización y manipulación de datos): ggplot2, dplyr, purrr,etc. |
| lubridate | Fácil manipulación de fechas y tiempos |
| ggrepel | Etiquetas sin superposición con ggplot2 |
#instalamos los paquetes si hace falta
if(!require("tidyverse")) install.packages("tidyverse")
if(!require("ggrepel")) install.packages("ggrepel")
if(!require("lubridate")) install.packages("lubridate")
#paquetes
library(tidyverse)
library(lubridate)
library(ggrepel)Preparar los datos
Primero importamos la precipitación y temperatura diaria de la estación meteorológica seleccionada (descarga). Usaremos los datos de Tenerife Sur (España) [1981-2020] accesible a través de Open Data AEMET. En R existe un paquete meteoland que facilita la descarga con funciones específicas para acceder a los datos de AEMET, Meteogalicia y Meteocat.
Paso 1: importar los datos
Importamos los datos en formato csv, siendo la primera columna la fecha, la segunda la precipitación (pr) y la última la temperatura media diaria (ta).
data <- read_csv("meteo_tenerife.csv") ## Rows: 14303 Columns: 3
## -- Column specification --------------------------------------------------------
## Delimiter: ","
## dbl (2): pr, ta
## date (1): date
##
## i Use `spec()` to retrieve the full column specification for this data.
## i Specify the column types or set `show_col_types = FALSE` to quiet this message.data## # A tibble: 14,303 x 3
## date pr ta
## <date> <dbl> <dbl>
## 1 1981-01-02 0 17.6
## 2 1981-01-03 0 16.8
## 3 1981-01-04 0 17.4
## 4 1981-01-05 0 17.6
## 5 1981-01-06 0 17
## 6 1981-01-07 0 17.6
## 7 1981-01-08 0 18.6
## 8 1981-01-09 0 19.8
## 9 1981-01-10 0 21.5
## 10 1981-01-11 3.8 17.6
## # ... with 14,293 more rowsPaso 2: preparar los datos
En el segundo paso preparamos los datos para calcular las anomalías. Para ello, creamos tres nuevas columnas: el mes, el año y la estación del año. Como nuestro objetivo son las anomalías invernales no podemos usar el año natural, ya que el invierno comprende el mes de diciembre de un año y los meses de enero y febrero del siguiente. La función personalizada meteo_yr() extrae el año de una fecha indicando el mes inicial. El concepto es similar al año hidrológico en el que se empieza en octubre.
meteo_yr <- function(dates, start_month = NULL) {
# convertir a POSIXlt
dates.posix <- as.POSIXlt(dates)
# la compensación
offset <- ifelse(dates.posix$mon >= start_month - 1, 1, 0)
# nuevo año
adj.year = dates.posix$year + 1900 + offset
return(adj.year)
}Usaremos las funciones de la colección de paquetes tidyverse (https://www.tidyverse.org/). La función mutate() ayuda a añadir nuevas columnas o a cambiar otras existentes. Para definir las estaciones del año, empleamos la función case_when() del paquete dplyr lo que tiene muchas ventajas en comparación con una cadena de ifelse(). En la función case_when() usamos fórmulas en dos tiempos, por un lado la condición y por otro la acción cuando se cumpla esa condición. En R una fórmula de dos tiempos o dos lados se consistuye con el operador ~. El operador binario %in% nos permite filtrar varios valores en un conjunto.
data <- mutate(data,
winter_yr = meteo_yr(date, 12),
month = month(date),
season = case_when(month %in% c(12,1:2) ~ "Winter",
month %in% 3:5 ~ "Spring",
month %in% 6:8 ~ "Summer",
month %in% 9:11 ~ "Autum"))
data## # A tibble: 14,303 x 6
## date pr ta winter_yr month season
## <date> <dbl> <dbl> <dbl> <dbl> <chr>
## 1 1981-01-02 0 17.6 1981 1 Winter
## 2 1981-01-03 0 16.8 1981 1 Winter
## 3 1981-01-04 0 17.4 1981 1 Winter
## 4 1981-01-05 0 17.6 1981 1 Winter
## 5 1981-01-06 0 17 1981 1 Winter
## 6 1981-01-07 0 17.6 1981 1 Winter
## 7 1981-01-08 0 18.6 1981 1 Winter
## 8 1981-01-09 0 19.8 1981 1 Winter
## 9 1981-01-10 0 21.5 1981 1 Winter
## 10 1981-01-11 3.8 17.6 1981 1 Winter
## # ... with 14,293 more rowsPaso 3: estimar las anomalías invernales
En el siguiente paso creamos un subset del invierno. Después agrupamos por el año meteorológico y calculamos la suma y el promedio para la precipitación y la temperatura, respectivamente. Para facilitar el trabajo el paquete magrittr introduce el operador llamado pipe en la forma %>% con el objetivo de combinar varias funciones sin la necesidad de asignar el resultado a un nuevo objeto. El operador pipe pasa la salida de una función aplicada al primer argumento de la siguiente función. Esta forma de combinar funciones permite encadenar varios pasos de forma simultánea. Se debe entender y pronunciar el %>% como “luego” (then).
data_inv <- filter(data,
season == "Winter") %>%
group_by(winter_yr) %>%
summarise(pr = sum(pr, na.rm = TRUE),
ta = mean(ta, na.rm = TRUE))Sólo nos quedan por calcular las anomalías de precipitación y temperatura. Las columnas pr_mean y ta_mean contendrán el promedio climatico, la referencia de las anomalías respecto al periodo normal 1981-2010. Por eso debemos filtrar los valores al periodo antes de 2010, lo que haremos de la forma habitual de filtrado de vectores en R. Una vez que tenemos las referencias estimamos las anomalías pr_anom y ta_anom. Para facilitar la interpretación, en el caso de la precipitación lo expresamos en porcentaje, pero poniendo el promedio en 0% en lugar del 100%.
Además, añadimmos tres columnas con información necesaria en la creación del gráfico. 1) labyr contiene el año de cada anomalía siempre y cuando haya sido mayor/menor del -+10% o -+0,5ºC, respectivamente (lo hago para que no haya demasiadas etiquetas), 2) symb_point una variable dummy para poder crear un simbolo diferencial entre los casos de (1), y 3) lab_font destacaremos en negrita el año 2020.
data_inv <- mutate(data_inv, pr_mean = mean(pr[winter_yr <= 2010]),
ta_mean = mean(ta[winter_yr <= 2010]),
pr_anom = (pr*100/pr_mean)-100,
ta_anom = ta-ta_mean,
labyr = case_when(pr_anom < -10 & ta_anom < -.5 ~ winter_yr,
pr_anom < -10 & ta_anom > .5 ~ winter_yr,
pr_anom > 10 & ta_anom < -.5 ~ winter_yr,
pr_anom > 10 & ta_anom > .5 ~ winter_yr),
symb_point = ifelse(!is.na(labyr), "yes", "no"),
lab_font = ifelse(labyr == 2020, "bold", "plain")
)Crear el gráfico
El gráfico lo construiremos añadiendo capa por capa los diferentes elementos: 1) el fondo con las diferentes cuadrículas (Seco-Cálido, Seco-Frío, etc.) 2) los puntos y etiquetas, y 3) los últimos ajustes de estilo.
Parte 1
La idea es que tengamos los puntos con anomalías seco-cálido en el cuadrante I (arriba-derecha) y los de húmedo-frío en el cuadrante III (abajo-izquierda). Por eso, debemos invertir el signo en las anomalías de precipitación. Después creamos un data.frame con las posiciones de las etiquetas de los cuatro cuadrantes. Para las posiciones en x y y se usan Inf y -Inf lo que equivale al punto máximo dentro del panel. No obstante, es necesario ajustar la posición hacia los puntos extremos dentro del marco gráfico con los argumentos conocidos de ggplot2: hjust y vjust.
data_inv_p <- mutate(data_inv, pr_anom = pr_anom * -1)
bglab <- data.frame(x = c(-Inf, Inf, -Inf, Inf),
y = c(Inf, Inf, -Inf, -Inf),
hjust = c(1, 1, 0, 0),
vjust = c(1, 0, 1, 0),
lab = c("Húmedo-Cálido", "Seco-Cálido",
"Húmedo-Frío", "Seco-Frío"))
bglab## x y hjust vjust lab
## 1 -Inf Inf 1 1 Húmedo-Cálido
## 2 Inf Inf 1 0 Seco-Cálido
## 3 -Inf -Inf 0 1 Húmedo-Frío
## 4 Inf -Inf 0 0 Seco-FríoParte 2
En la segunda podemos empezar a construir el gráfico añadiendo todos los elementos gráficos. En esta parte creamos el fondo con los diferentes colores de cada cuadrante. La función annotate() permite añadir capas de geometría sin el uso de variables dentro de un data.frame. Con la función geom_hline() y geom_vline() marcamos los cuadrantes en horizontal y vertical usando una linea discontinua. Por último, dibujamos las etiquetas de cada cuadrante, empleando la función geom_text(). Cuando usamos diferentes fuentes de data.frames, uno diferente al principal usado en ggplot(), debemos indicarlo con el argumento data en la función de geomtría correspondiente.
g1 <- ggplot(data_inv_p,
aes(pr_anom, ta_anom)) +
annotate("rect", xmin = -Inf, xmax = 0, ymin = 0, ymax = Inf, fill = "#fc9272", alpha = .6) + #humedo-calido
annotate("rect", xmin = 0, xmax = Inf, ymin = 0, ymax = Inf, fill = "#cb181d", alpha = .6) + #seco-calido
annotate("rect", xmin = -Inf, xmax = 0, ymin = -Inf, ymax = 0, fill = "#2171b5", alpha = .6) + #humedo-frio
annotate("rect", xmin = 0, xmax = Inf, ymin = -Inf, ymax = 0, fill = "#c6dbef", alpha = .6) + #seco-frio
geom_hline(yintercept = 0,
linetype = "dashed") +
geom_vline(xintercept = 0,
linetype = "dashed") +
geom_text(data = bglab,
aes(x, y, label = lab, hjust = hjust, vjust = vjust),
fontface = "italic", size = 5,
angle = 90, colour = "white")
g1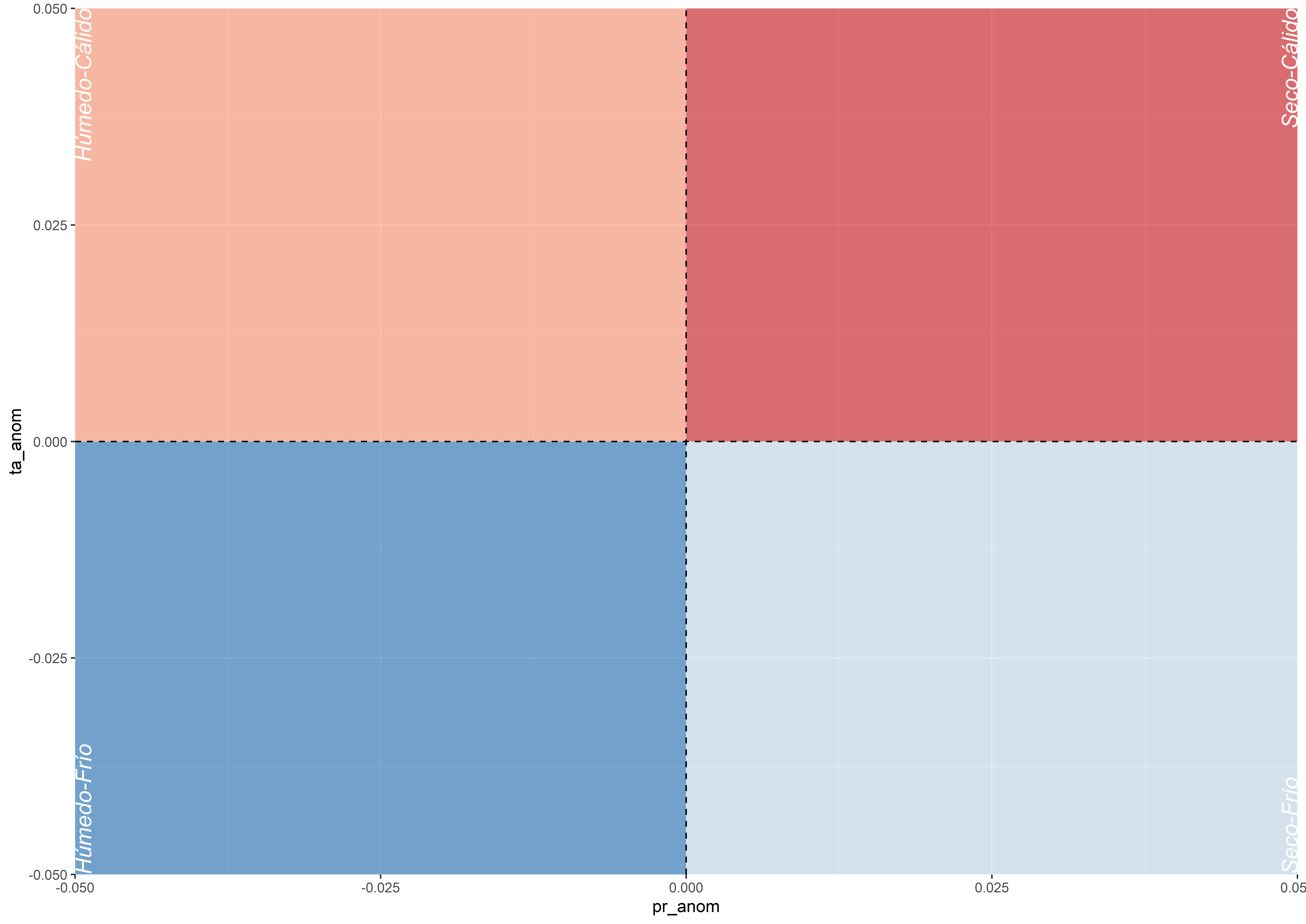
Parte 3
En la tercera parte simplemente añadimos los puntos de las anomalías y las etiquetas de los años. La función geom_text_repel() es similar a la que conocemos por defecto en ggplot2, geom_text(), pero evita el sobrlapso entre sí.
g2 <- g1 + geom_point(aes(fill = symb_point, colour = symb_point),
size = 2.8, shape = 21, show.legend = FALSE) +
geom_text_repel(aes(label = labyr, fontface = lab_font),
max.iter = 5000,
size = 3.5)
g2## Warning: Removed 25 rows containing missing values (geom_text_repel).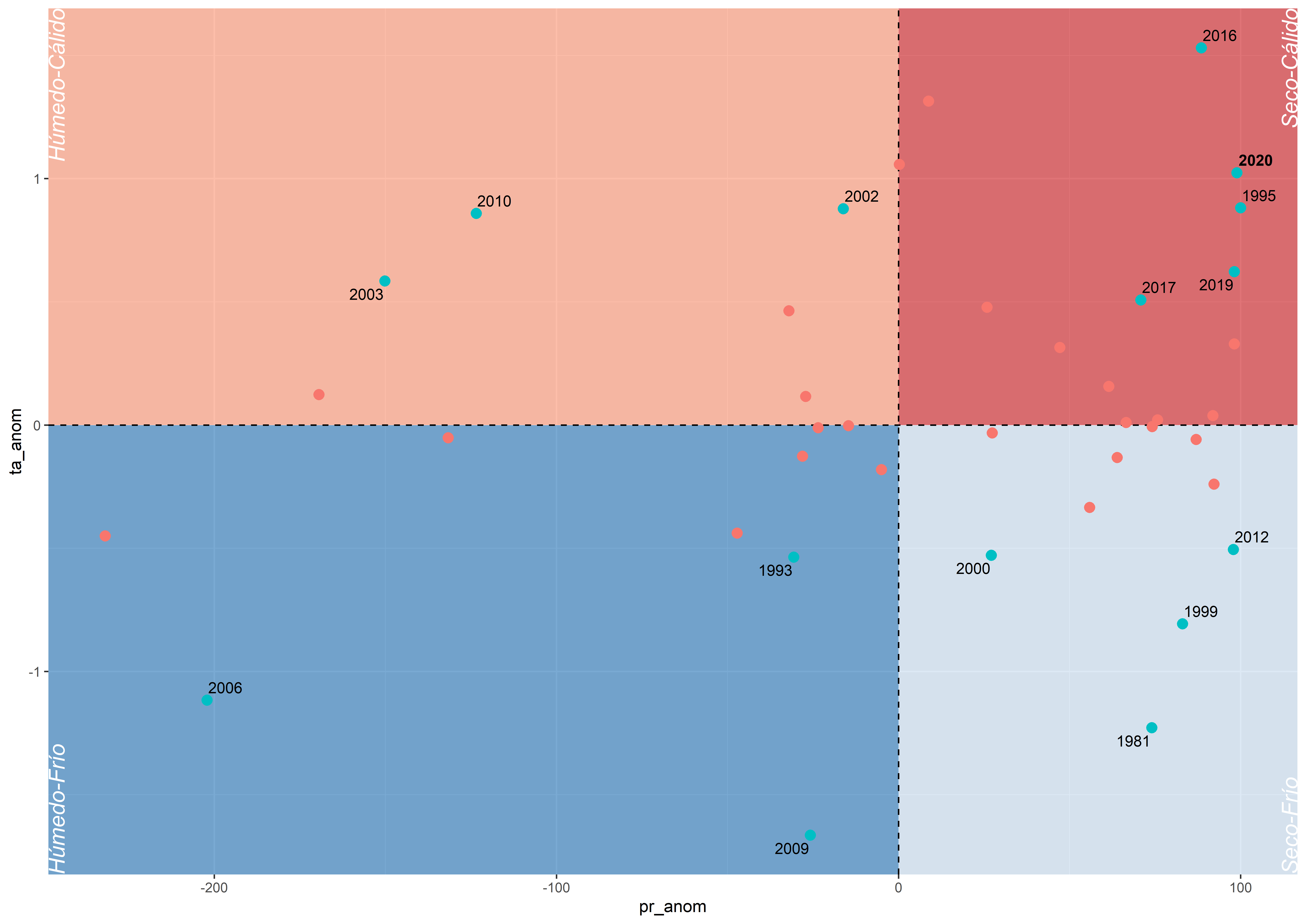
Parte 4
En la última parte ajustamos, además del estilo general, los ejes, el tipo de color y el (sub)título. Recuerda que cambiamos el signo en las anomalías de precipitación. Por eso, debemos usar los argumentos breaks y labels en la función scale_x_continouous() para revertir el signo en las etiquetas correspondientes a los cortes.
g3 <- g2 + scale_x_continuous("Anomalía de precipitación en %",
breaks = seq(-100, 250, 10) * -1,
labels = seq(-100, 250, 10),
limits = c(min(data_inv_p$pr_anom), 100)) +
scale_y_continuous("Anomalía de temperatura media en ºC",
breaks = seq(-2, 2, 0.5)) +
scale_fill_manual(values = c("black", "white")) +
scale_colour_manual(values = rev(c("black", "white"))) +
labs(title = "Anomalías invernales en Tenerife Sur",
caption = "Datos: AEMET\nPeriodo normal 1981-2010") +
theme_bw()
g3## Warning: Removed 25 rows containing missing values (geom_text_repel).