Una muy breve introducción a Tidyverse
1 Tidyverse
El universo de los paquetes de tidyverse, una colección de paquetes de funciones para un uso especialmente enfocado en la ciencia de datos, abrió un antes y después en la programación de R. En este post voy a resumir muy brevemente lo más esencial para inicarse en este mundo. La gramática sigue en todas las funciones una estructura común. Lo más esencial es que el primer argumento es el objeto y a continuación viene el resto de argumentos. Además, se proporciona un conjunto de verbos que facilitan el uso de las funciones. En la actualidad, la filosofía de las funciones también se refleja en otros paquetes que hacen compatible su uso con la colección de tidyverse. Por ejemplo, el paquete sf (simple feature) para el tratamiento de datos vectoriales, permite el uso de múltiples funciones que encontramos en el paquete dplyr.
El núcleo de la colección lo constituyen los siguientes paquetes:
| Paquete | Descripción |
|---|---|
| ggplot2 | Gramática para la creación de gráficos |
| purrr | Programación funcional de R |
| tibble | Sistema moderno y efectivo de tablas |
| dplyr | Gramatica para la manipulación de datos |
| tidyr | Conjunto de funciones para ordenar datos |
| stringr | Conjunto de funciones para trabajar con caracteres |
| readr | Una forma fácil y rápida para importar datos |
| forcats | Herramientas y funciones para trabajar fácilmente con factores |
Además de los paquetes menciondos, también se usa muy frecuentemente lubridate para trabajar con fechas y horas, y también readxl que nos permite importar archivos en formato Excel. Para conocer todos los paquetes disponibles podemos emplear la función tidyverse_packages().
## [1] "broom" "cli" "crayon" "dbplyr"
## [5] "dplyr" "dtplyr" "forcats" "googledrive"
## [9] "googlesheets4" "ggplot2" "haven" "hms"
## [13] "httr" "jsonlite" "lubridate" "magrittr"
## [17] "modelr" "pillar" "purrr" "readr"
## [21] "readxl" "reprex" "rlang" "rstudioapi"
## [25] "rvest" "stringr" "tibble" "tidyr"
## [29] "xml2" "tidyverse"Es muy fácil encontrarnos con conflicos de funciones, o sea, que el mismo nombre de función exista en varios paquetes. Para evitarlo, podemos escribir el nombre del paquete delante de la función que queremos usar, separados por el símbolo de dos puntos escrito dos veces (package_name::function_name).
Antes de empezar con los paquetes, espero que sea verdaderamente una breve introducción, algunos comentarios sobre el estilo al programar en R.
2 Guía de estilo
En R no existe una guía de estilo universal, o sea, en la sintaxis de R no es necesario seguir normas concretas para nuestros scripts. Es recomendable trabajar de forma homogénea y clara a la hora de escribir con un estilo uniforme y legible. La colección de tidyverse tiene una guia propia (https://style.tidyverse.org/).
Las recomendaciones más importes son:
- Evitar usar más de 80 caracteres por línea para permitir leer el código completo.
- Usar siempre un espacio después de una coma, nunca antes.
- Los operadores (==, +, -, <-, %>%, etc.) deben tener un espacio antes y después.
- No hay espacio entre el nombre de una función y el primer paréntesis, ni entre el último arguemento y el paréntesis final de una función.
- Evitar reutilizar nombres de funciones y variables comunes (
c <- 5vs.c()) - Ordenar el script separando las partes con la forma de comentario
# Importar datos ----- - Se deben evitar tildes o símbolos especiales en nombres, archivos, rutas, etc.
- Nombres de los objetos deben seguir una estructura constante:
day_one,day_1.
Es aconsejable usar una correcta indentación para multiples argumentos de una función o funciones encadenadas por el operador pipe (%>%).
3 Pipe %>%
Para facilitar el trabajo en la gestión, manipulación y visualización de datos, el paquete magrittr introduce el operador llamado pipe en la forma %>% con el objetivo de combinar varias funciones sin la necesidad de asignar el resultado a un nuevo objeto. El operador pipe pasa a la salida de una función aplicada al primer argumento de la siguiente función. Esta forma de combinar funciones permite encadenar varios pasos de forma simultánea. En el siguiente ejemplo, muy sencillo, pasamos el vector 1:5 a la función mean() para calcular el promedio.
1:5 %>% mean()## [1] 34 Paquetes de Tidyverse
4.1 Lectura y escritura
El paquete readr facilita la lectura o escritura de múltiples formatos de archivo usando funciones que comienzan por read_* o write_*.
En comparación con R Base las funciones son más rápidas, ayudan a limpiar los nombres de las columnas y las fechas son convertidas automáticamente. Las tablas importadas son de clase tibble (tbl_df), una versión moderna de data.frame del paquete tibble. En el mismo sentido se puede usar la función read_excel() del paquete readxl para importar datos de hojas de Excel (más detalles también en esta entrada de mi blog). En el siguiente ejemplo importamos los datos de la movilidad registrada por Google (enlace) durante los últimos meses a causa de la pandemia COVID-19 (descarga).
| Función lectura | Descripción |
|---|---|
| read_csv() o read_csv2() | coma o punto-coma (CSV) |
| read_delim() | separador general |
| read_table() | espacio blanco |
# cargar el paquete
library(tidyverse)
google_mobility <- read_csv("Global_Mobility_Report.csv")## Rows: 516697 Columns: 13
## -- Column specification --------------------------------------------------------
## Delimiter: ","
## chr (6): country_region_code, country_region, sub_region_1, sub_region_2, i...
## dbl (6): retail_and_recreation_percent_change_from_baseline, grocery_and_ph...
## date (1): date
##
## i Use `spec()` to retrieve the full column specification for this data.
## i Specify the column types or set `show_col_types = FALSE` to quiet this message.google_mobility## # A tibble: 516,697 x 13
## country_region_code country_region sub_region_1 sub_region_2 iso_3166_2_code
## <chr> <chr> <chr> <chr> <chr>
## 1 AE United Arab Em~ <NA> <NA> <NA>
## 2 AE United Arab Em~ <NA> <NA> <NA>
## 3 AE United Arab Em~ <NA> <NA> <NA>
## 4 AE United Arab Em~ <NA> <NA> <NA>
## 5 AE United Arab Em~ <NA> <NA> <NA>
## 6 AE United Arab Em~ <NA> <NA> <NA>
## 7 AE United Arab Em~ <NA> <NA> <NA>
## 8 AE United Arab Em~ <NA> <NA> <NA>
## 9 AE United Arab Em~ <NA> <NA> <NA>
## 10 AE United Arab Em~ <NA> <NA> <NA>
## # ... with 516,687 more rows, and 8 more variables: census_fips_code <chr>,
## # date <date>, retail_and_recreation_percent_change_from_baseline <dbl>,
## # grocery_and_pharmacy_percent_change_from_baseline <dbl>,
## # parks_percent_change_from_baseline <dbl>,
## # transit_stations_percent_change_from_baseline <dbl>,
## # workplaces_percent_change_from_baseline <dbl>,
## # residential_percent_change_from_baseline <dbl>Debemos prestar atención a los nombres de los argumentos, ya que cambian en las funciones de readr. Por ejemplo, el argumento conocido header = TRUE de read.csv() es en este caso col_names = TRUE. Podemos encontrar más detalles en el Cheat-Sheet de readr .
4.2 Manipulación de caracteres
Cuando se requiere manipular cadenas de texto usamos el paquete stringr, cuyas funciones siempre empiezan por str_* seguidas por un verbo y el primer argumento.
Algunas de estas funciones son las siguientes:
| Función | Descripción |
|---|---|
| str_replace() | reemplazar patrones |
| str_c() | combinar characteres |
| str_detect() | detectar patrones |
| str_extract() | extraer patrones |
| str_sub() | extraer por posición |
| str_length() | longitud de la cadena de caracteres |
Se suelen usar expresiones regulares para patrones de caracteres. Por ejemplo, la expresión regular [aeiou] coincide con cualquier caracter único que sea una vocal. El uso de corchetes [] corresponde a clases de caracteres. Por ejemplo, [abc] corresponde a cada letra independientemente de la posición. [a-z] o [A-Z] o [0-9] cada uno entre a y z ó 0 y 9. Y por último, [:punct:] puntuación, etc. Con llaves “{}” podemos indicar el número del elemento anterior {2} sería dos veces, {1,2} entre una y dos, etc. Además con $o ^ podemos indicar si el patrón empieza al principio o termina al final. Podemos encontrar más detalles y patrones en el Cheat-Sheet de stringr.
# reemplazamos 'er' al final por vacío
str_replace(month.name, "er$", "")## [1] "January" "February" "March" "April" "May" "June"
## [7] "July" "August" "Septemb" "Octob" "Novemb" "Decemb"str_replace(month.name, "^Ma", "")## [1] "January" "February" "rch" "April" "y" "June"
## [7] "July" "August" "September" "October" "November" "December"# combinar caracteres
a <- str_c(month.name, 1:12, sep = "_")
a## [1] "January_1" "February_2" "March_3" "April_4" "May_5"
## [6] "June_6" "July_7" "August_8" "September_9" "October_10"
## [11] "November_11" "December_12"# colapsar combinación
str_c(month.name, collapse = ", ")## [1] "January, February, March, April, May, June, July, August, September, October, November, December"# dedectamos patrones
str_detect(a, "_[1-5]{1}")## [1] TRUE TRUE TRUE TRUE TRUE FALSE FALSE FALSE FALSE TRUE TRUE TRUE# extraemos patrones
str_extract(a, "_[1-9]{1,2}")## [1] "_1" "_2" "_3" "_4" "_5" "_6" "_7" "_8" "_9" "_1" "_11" "_12"# extraermos los caracteres en las posiciones entre 1 y 2
str_sub(month.name, 1, 2)## [1] "Ja" "Fe" "Ma" "Ap" "Ma" "Ju" "Ju" "Au" "Se" "Oc" "No" "De"# longitud de cada mes
str_length(month.name)## [1] 7 8 5 5 3 4 4 6 9 7 8 8# con pipe, el '.' representa al objeto que pasa el operador %>%
str_length(month.name) %>%
str_c(month.name, ., sep = ".")## [1] "January.7" "February.8" "March.5" "April.5" "May.3"
## [6] "June.4" "July.4" "August.6" "September.9" "October.7"
## [11] "November.8" "December.8"Una función muy útil es str_glue() para interpolar caracteres.
name <- c("Juan", "Michael")
age <- c(50, 80)
date_today <- Sys.Date()
str_glue(
"My name is {name}, ",
"I'am {age}, ",
"and my birth year is {format(date_today-age*365, '%Y')}."
)## My name is Juan, I'am 50, and my birth year is 1972.
## My name is Michael, I'am 80, and my birth year is 1942.4.3 Manejo de fechas y horas
El paquete lubridate ayuda en el manejo de fechas y horas. Nos permite crear los objetos reconocidos por R con funciones (como ymd() ó ymd_hms()) y hacer cálculos.
Debemos conocer las siguientes abreviaturas:
ymd: representay:year,m:month,d:dayhms: representah:hour,m:minutes,s:seconds
# paquete
library(lubridate)##
## Attaching package: 'lubridate'## The following objects are masked from 'package:base':
##
## date, intersect, setdiff, union# vector de fechas
dat <- c("1999/12/31", "2000/01/07", "2005/05/20","2010/03/25")
# vector de fechas y horas
dat_time <- c("1988-08-01 05:00", "2000-02-01 22:00")
# convertir a clase date
dat <- ymd(dat)
dat## [1] "1999-12-31" "2000-01-07" "2005-05-20" "2010-03-25"# otras formatos
dmy("05-02-2000")## [1] "2000-02-05"ymd("20000506")## [1] "2000-05-06"# convertir a POSIXct
dat_time <- ymd_hm(dat_time)
dat_time## [1] "1988-08-01 05:00:00 UTC" "2000-02-01 22:00:00 UTC"# diferentes formatos en un vector
dat_mix <- c("1999/12/05", "05-09-2008", "2000/08/09", "25-10-2019")
# indicar formato con la convención conocida en ?strptime
parse_date_time(dat_mix, order = c("%Y/%m/%d", "%d-%m-%Y"))## [1] "1999-12-05 UTC" "2008-09-05 UTC" "2000-08-09 UTC" "2019-10-25 UTC"Más funciones útiles:
# extraer el año
year(dat)## [1] 1999 2000 2005 2010# el mes
month(dat)## [1] 12 1 5 3month(dat, label = TRUE) # como etiqueta## [1] dic ene may mar
## 12 Levels: ene < feb < mar < abr < may < jun < jul < ago < sep < ... < dic# el día de la semana
wday(dat)## [1] 6 6 6 5wday(dat, label = TRUE) # como etiqueta## [1] vi\\. vi\\. vi\\. ju\\.
## Levels: do\\. < lu\\. < ma\\. < mi\\. < ju\\. < vi\\. < sá\\.# la hora
hour(dat_time)## [1] 5 22# sumar 10 días
dat + days(10)## [1] "2000-01-10" "2000-01-17" "2005-05-30" "2010-04-04"# sumar 1 mes
dat + months(1)## [1] "2000-01-31" "2000-02-07" "2005-06-20" "2010-04-25"Por último, la función make_date() es muy útil en crear fechas a partir de diferentes partes de las mismas como puede ser el año, mes, etc.
# crear fecha a partir de sus elementos, aquí con año y mes
make_date(2000, 5)## [1] "2000-05-01"# crear fecha con hora
make_datetime(2005, 5, 23, 5)## [1] "2005-05-23 05:00:00 UTC"Podemos encontrar más detalles en el Cheat-Sheet de lubridate.
4.4 Manipulación de tablas y vectores
Los paquetes dplyr y tidyr nos proporciona una gramática de manipulación de datos con un conjunto de verbos útiles para resolver los problemas más comunes. Las funciones más importantes son:
| Función | Descripción |
|---|---|
| mutate() | añadir nuevas variables o modificar existentes |
| select() | seleccionar variables |
| filter() | filtrar |
| summarise() | resumir/reducir |
| arrange() | ordenar |
| group_by() | agrupar |
| rename() | renombrar columnas |
En caso de que no lo hayas hecho antes, importamos los datos de movilidad.
google_mobility <- read_csv("Global_Mobility_Report.csv")## Rows: 516697 Columns: 13
## -- Column specification --------------------------------------------------------
## Delimiter: ","
## chr (6): country_region_code, country_region, sub_region_1, sub_region_2, i...
## dbl (6): retail_and_recreation_percent_change_from_baseline, grocery_and_ph...
## date (1): date
##
## i Use `spec()` to retrieve the full column specification for this data.
## i Specify the column types or set `show_col_types = FALSE` to quiet this message.4.4.1 Selecionar y renombrar
Podemos selecionar o eliminar columnas con la función select(), usando el nombre o índice de la(s) columna(s). Para suprimir columnas hacemos uso del signo negativo. La función rename ayuda en renombrar columnas o bien con el mismo nombre o con su índice.
residential_mobility <- select(google_mobility,
country_region_code:sub_region_1,
date,
residential_percent_change_from_baseline) %>%
rename(resi = 5)4.4.2 Filtrar y ordenar
Para filtrar datos, empleamos filter() con operadores lógicos (|, ==, >, etc) o funciones que devuelven un valor lógico (str_detect(), is.na(), etc.). La función arrange() ordena de menor a mayor por una o múltiples variables (con el signo negativo - se invierte el orden de mayor a menor).
filter(residential_mobility,
country_region_code == "US")## # A tibble: 304,648 x 5
## country_region_code country_region sub_region_1 date resi
## <chr> <chr> <chr> <date> <dbl>
## 1 US United States <NA> 2020-02-15 -1
## 2 US United States <NA> 2020-02-16 -1
## 3 US United States <NA> 2020-02-17 5
## 4 US United States <NA> 2020-02-18 1
## 5 US United States <NA> 2020-02-19 0
## 6 US United States <NA> 2020-02-20 1
## 7 US United States <NA> 2020-02-21 0
## 8 US United States <NA> 2020-02-22 -1
## 9 US United States <NA> 2020-02-23 -1
## 10 US United States <NA> 2020-02-24 0
## # ... with 304,638 more rowsfilter(residential_mobility,
country_region_code == "US",
sub_region_1 == "New York")## # A tibble: 7,068 x 5
## country_region_code country_region sub_region_1 date resi
## <chr> <chr> <chr> <date> <dbl>
## 1 US United States New York 2020-02-15 0
## 2 US United States New York 2020-02-16 -1
## 3 US United States New York 2020-02-17 9
## 4 US United States New York 2020-02-18 3
## 5 US United States New York 2020-02-19 2
## 6 US United States New York 2020-02-20 2
## 7 US United States New York 2020-02-21 3
## 8 US United States New York 2020-02-22 -1
## 9 US United States New York 2020-02-23 -1
## 10 US United States New York 2020-02-24 0
## # ... with 7,058 more rowsfilter(residential_mobility,
resi > 50) %>%
arrange(-resi)## # A tibble: 32 x 5
## country_region_code country_region sub_region_1 date resi
## <chr> <chr> <chr> <date> <dbl>
## 1 KW Kuwait Al Farwaniyah Governorate 2020-05-14 56
## 2 KW Kuwait Al Farwaniyah Governorate 2020-05-21 55
## 3 SG Singapore <NA> 2020-05-01 55
## 4 KW Kuwait Al Farwaniyah Governorate 2020-05-28 54
## 5 PE Peru Metropolitan Municipalit~ 2020-04-10 54
## 6 EC Ecuador Pichincha 2020-03-27 53
## 7 KW Kuwait Al Farwaniyah Governorate 2020-05-11 53
## 8 KW Kuwait Al Farwaniyah Governorate 2020-05-13 53
## 9 KW Kuwait Al Farwaniyah Governorate 2020-05-20 53
## 10 SG Singapore <NA> 2020-04-10 53
## # ... with 22 more rows4.4.3 Agrupar y resumir
¿Dónde encontramos mayor variabilidad entre regiones en cada país el día 1 de abril de 2020?
Para responder a esta pregunta, primero filtramos los datos y después agrupamos por la columna de país. Cuando empleamos la función summarise() posterior a la agrupación, nos permite resumir por estos grupos. Incluso, la combinación del group_by() con la función mutate() permite modificar columnas por grupos. En summarise() calculamos el valor máximo, mínimo y la diferencia entre ambos extremos creando nuevas columnas.
resi_variability <- residential_mobility %>%
filter(date == ymd("2020-04-01"),
!is.na(sub_region_1)) %>%
group_by(country_region) %>%
summarise(mx = max(resi, na.rm = TRUE),
min = min(resi, na.rm = TRUE),
range = abs(mx)-abs(min))## Warning in max(resi, na.rm = TRUE): ningun argumento finito para max; retornando
## -Inf
## Warning in max(resi, na.rm = TRUE): ningun argumento finito para max; retornando
## -Inf
## Warning in max(resi, na.rm = TRUE): ningun argumento finito para max; retornando
## -Inf
## Warning in max(resi, na.rm = TRUE): ningun argumento finito para max; retornando
## -Inf
## Warning in max(resi, na.rm = TRUE): ningun argumento finito para max; retornando
## -Inf
## Warning in max(resi, na.rm = TRUE): ningun argumento finito para max; retornando
## -Inf## Warning in min(resi, na.rm = TRUE): ningún argumento finito para min; retornando
## Inf
## Warning in min(resi, na.rm = TRUE): ningún argumento finito para min; retornando
## Inf
## Warning in min(resi, na.rm = TRUE): ningún argumento finito para min; retornando
## Inf
## Warning in min(resi, na.rm = TRUE): ningún argumento finito para min; retornando
## Inf
## Warning in min(resi, na.rm = TRUE): ningún argumento finito para min; retornando
## Inf
## Warning in min(resi, na.rm = TRUE): ningún argumento finito para min; retornando
## Infarrange(resi_variability, -range)## # A tibble: 94 x 4
## country_region mx min range
## <chr> <dbl> <dbl> <dbl>
## 1 Nigeria 43 6 37
## 2 United States 35 6 29
## 3 India 36 15 21
## 4 Malaysia 45 26 19
## 5 Philippines 40 21 19
## 6 Vietnam 28 9 19
## 7 Colombia 41 24 17
## 8 Ecuador 44 27 17
## 9 Argentina 35 19 16
## 10 Chile 30 14 16
## # ... with 84 more rows4.4.4 Unir tablas
¿Cómo podemos filtrar los datos para obtener un subconjunto de Europa?
Para ello, importamos datos espaciales con el código de país y una columna de las regiones. Explicaciones detalladas sobre el paquete sf (simple feature) para trabajar con datos vectoriales, lo dejaremos para otro post.
library(rnaturalearth) # paquete de datos vectoriales
# datos de países
wld <- ne_countries(returnclass = "sf")
# filtramos los países con código y seleccionamos las dos columnas de interés
wld <- filter(wld, !is.na(iso_a2)) %>% select(iso_a2, subregion)
# plot
plot(wld)
Otras funciones de dplyr nos permiten unir tablas: *_join(). Según hacia qué tabla (izquierda o derecha) se quiere unir, cambia la función : left_join(), right_join() o incluso full_join(). El argumento by no es necesario siempre y cuando ambas tablas tienen una columna en común. No obstante, en este caso la columna de fusión es diferente, por eso, usamos el modo c("country_region_code"="iso_a2"). El paquete forcats de tidyverse tiene muchas funciones útiles para manejar variables categóricas (factors), variables que tienen un conjunto fijo y conocido de valores posibles. Todas las funciones de forcats tienen el prefijo fct_*. Por ejemplo, en este caso usamos fct_reorder() para reordenar las etiquetas de los países en orden de la máxima basada en los registros de movibilidad residencial. Finalmente, creamos una nueva columna ‘resi_real’ para cambiar el valor de referencia, el promedio (baseline), fijado en 0 a 100.
subset_europe <- filter(residential_mobility,
is.na(sub_region_1),
!is.na(resi)) %>%
left_join(wld, by = c("country_region_code"="iso_a2")) %>%
filter(subregion %in% c("Northern Europe",
"Southern Europe",
"Western Europe",
"Eastern Europe")) %>%
mutate(resi_real = resi + 100,
region = fct_reorder(country_region,
resi,
.fun = "max",
.desc = FALSE)) %>%
select(-geometry, -sub_region_1)
str(subset_europe)## tibble [3,988 x 7] (S3: tbl_df/tbl/data.frame)
## $ country_region_code: chr [1:3988] "AT" "AT" "AT" "AT" ...
## $ country_region : chr [1:3988] "Austria" "Austria" "Austria" "Austria" ...
## $ date : Date[1:3988], format: "2020-02-15" "2020-02-16" ...
## $ resi : num [1:3988] -2 -2 0 0 1 0 1 -2 0 -1 ...
## $ subregion : chr [1:3988] "Western Europe" "Western Europe" "Western Europe" "Western Europe" ...
## $ resi_real : num [1:3988] 98 98 100 100 101 100 101 98 100 99 ...
## $ region : Factor w/ 35 levels "Belarus","Ukraine",..: 18 18 18 18 18 18 18 18 18 18 ...4.4.5 Tablas largas y anchas
Antes de pasar a la visualización con ggplot2. Es muy habitual modificar la tabla entre dos formatos principales. Una tabla es tidy cuando 1) cada variable es una columna 2) cada observación/caso es una fila y 3) cada tipo de unidad observacional forma una tabla.
# subconjunto
mobility_selection <- select(subset_europe, country_region_code, date:resi)
mobility_selection## # A tibble: 3,988 x 3
## country_region_code date resi
## <chr> <date> <dbl>
## 1 AT 2020-02-15 -2
## 2 AT 2020-02-16 -2
## 3 AT 2020-02-17 0
## 4 AT 2020-02-18 0
## 5 AT 2020-02-19 1
## 6 AT 2020-02-20 0
## 7 AT 2020-02-21 1
## 8 AT 2020-02-22 -2
## 9 AT 2020-02-23 0
## 10 AT 2020-02-24 -1
## # ... with 3,978 more rows# tabla ancha
mobi_wide <- pivot_wider(mobility_selection,
names_from = country_region_code,
values_from = resi)
mobi_wide## # A tibble: 114 x 36
## date AT BA BE BG BY CH CZ DE DK EE ES
## <date> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
## 1 2020-02-15 -2 -1 -1 0 -1 -1 -2 -1 0 0 -2
## 2 2020-02-16 -2 -1 1 -3 0 -1 -1 0 1 0 -2
## 3 2020-02-17 0 -1 0 -2 0 1 0 0 1 1 -1
## 4 2020-02-18 0 -1 0 -2 0 1 0 1 1 1 0
## 5 2020-02-19 1 -1 0 -1 -1 1 0 1 1 0 -1
## 6 2020-02-20 0 -1 0 0 -1 0 0 1 1 0 -1
## 7 2020-02-21 1 -2 0 -1 -1 1 0 2 1 1 -2
## 8 2020-02-22 -2 -1 0 0 -2 -2 -3 0 1 0 -2
## 9 2020-02-23 0 -1 0 -3 -1 -1 0 0 0 -2 -3
## 10 2020-02-24 -1 -1 4 -1 0 0 0 4 0 16 0
## # ... with 104 more rows, and 24 more variables: FI <dbl>, FR <dbl>, GB <dbl>,
## # GR <dbl>, HR <dbl>, HU <dbl>, IE <dbl>, IT <dbl>, LT <dbl>, LU <dbl>,
## # LV <dbl>, MD <dbl>, MK <dbl>, NL <dbl>, NO <dbl>, PL <dbl>, PT <dbl>,
## # RO <dbl>, RS <dbl>, RU <dbl>, SE <dbl>, SI <dbl>, SK <dbl>, UA <dbl># tabla larga
pivot_longer(mobi_wide,
2:36,
names_to = "country_code",
values_to = "resi")## # A tibble: 3,990 x 3
## date country_code resi
## <date> <chr> <dbl>
## 1 2020-02-15 AT -2
## 2 2020-02-15 BA -1
## 3 2020-02-15 BE -1
## 4 2020-02-15 BG 0
## 5 2020-02-15 BY -1
## 6 2020-02-15 CH -1
## 7 2020-02-15 CZ -2
## 8 2020-02-15 DE -1
## 9 2020-02-15 DK 0
## 10 2020-02-15 EE 0
## # ... with 3,980 more rowsOtro grupo de funciones a las que deberías echar un vistazo son: separate(), case_when(), complete(). Podemos encontrar más detalles en el Cheat-Sheet de dplyr
4.5 Visualizar datos
ggplot2 es un sistema moderno, y con una enorme variedad de opciones, para visualización de datos. A diferencia del sistema gráfico de R Base se utiliza una gramática diferente.
La gramática de los gráficos (grammar of graphics, de allí “gg”) consiste en la suma de varias capas u objetos independientes que se combinan usando + para construir el gráfico final. ggplot diferencia entre los datos, lo que se visualiza y la forma en que se visualiza.
data: nuestro conjunto de datos (
data.frameotibble)aesthetics: con la función
aes()indicamos las variables que corresponden a los ejes x, y, z,… o, cuando se pretende aplicar parámetros gráficos (color, size, shape) según una variable. Es posible incluiraes()enggplot()o en la función correspondiente a una geometríageom_*.geometries: son objetos
geom_*que indican la geometría a usar, (p. ej.:geom_point(),geom_line(),geom_boxplot(), etc.).scales: son objetos de tipo
scales_*(p. ej.:scale_x_continous(),scale_colour_manual()) para manipular las ejes, definir colores, etc.statistics: son objetos
stat_*(p.ej.:stat_density()) que permiten aplicar transformaciones estadísticas.
Podemos encontrar más detalles en el Cheat-Sheet de ggplot2. ggplot es complementado constantemente con extensiones para geometrías u otras opciones gráficas (https://exts.ggplot2.tidyverse.org/ggiraph.html), para obtener ideas gráficas, debes echarle un vistazo a la Galería de Gráficos R (https://www.r-graph-gallery.com/).
4.5.1 Gráfico de linea y puntos
Creamos un subconjunto de nuestros datos de movilidad para residencias y parques, filtrando los registros de regiones italianas. Además, dividimos los valores de movilidad en porcentaje por 100 para obtener la fracción, ya que ggplot2 nos permite indicar la unidad de porcentaje en el argumento de las etiquetas (último gráfico de esta sección).
# creamos el subconjunto
it <- filter(google_mobility,
country_region == "Italy",
is.na(sub_region_1)) %>%
mutate(resi = residential_percent_change_from_baseline/100,
parks = parks_percent_change_from_baseline/100)
# gráfico de línea
ggplot(it,
aes(date, resi)) +
geom_line()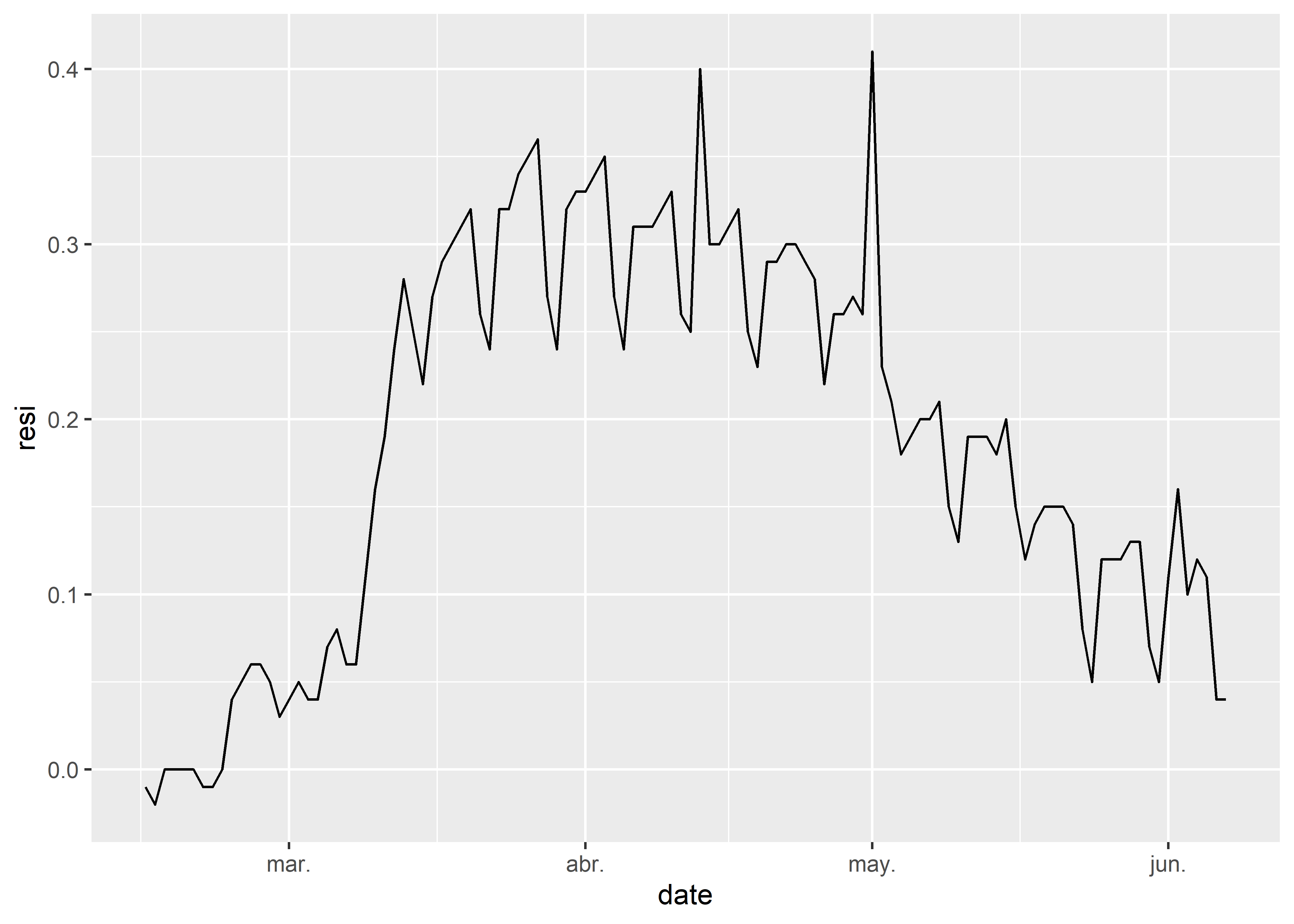
# gráfico de dispersión con línea de correlación
ggplot(it,
aes(parks, resi)) +
geom_point() +
geom_smooth(method = "lm")## `geom_smooth()` using formula 'y ~ x'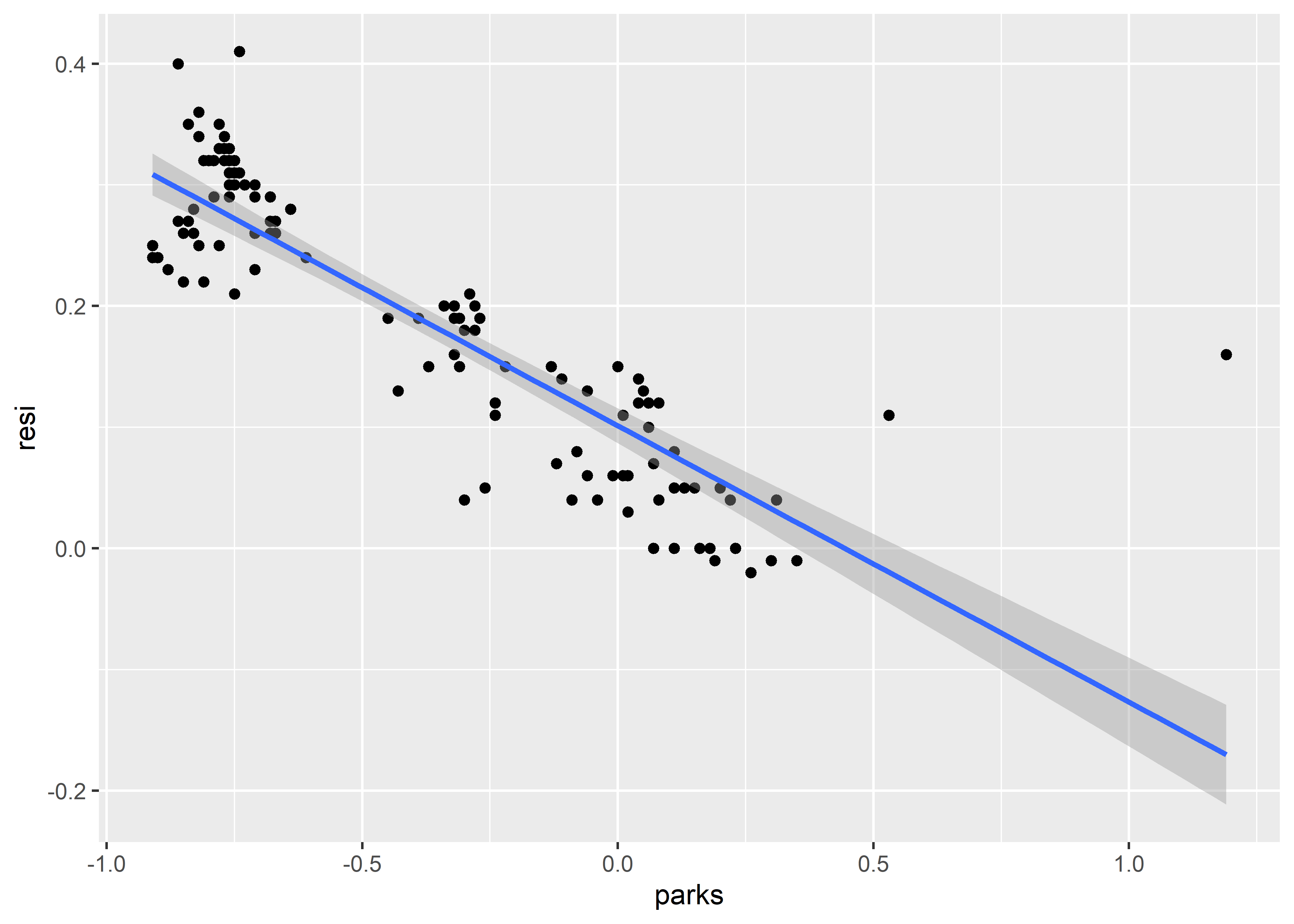
Para modificar los ejes, empleamos las diferentes funciones de scale_* que debemos adpatar a las escalas de medición (date, discrete, continuous, etc.). La función labs() nos ayuda en definir los títulos de ejes, del gráfico y de la leyenda. Por último, añadimos con theme_light() el estilo del gráfico (otros son theme_bw(), theme_minimal(), etc.). También podríamos hacer cambios de todos los elementos gráficos a través de theme().
# time serie plot
ggplot(it,
aes(date, resi)) +
geom_line(colour = "#560A86", size = 0.8) +
scale_x_date(date_breaks = "10 days",
date_labels = "%d %b") +
scale_y_continuous(breaks = seq(-0.1, 1, 0.1),
labels = scales::percent) +
labs(x = "",
y = "Residential mobility",
title = "Mobility during COVID-19") +
theme_light()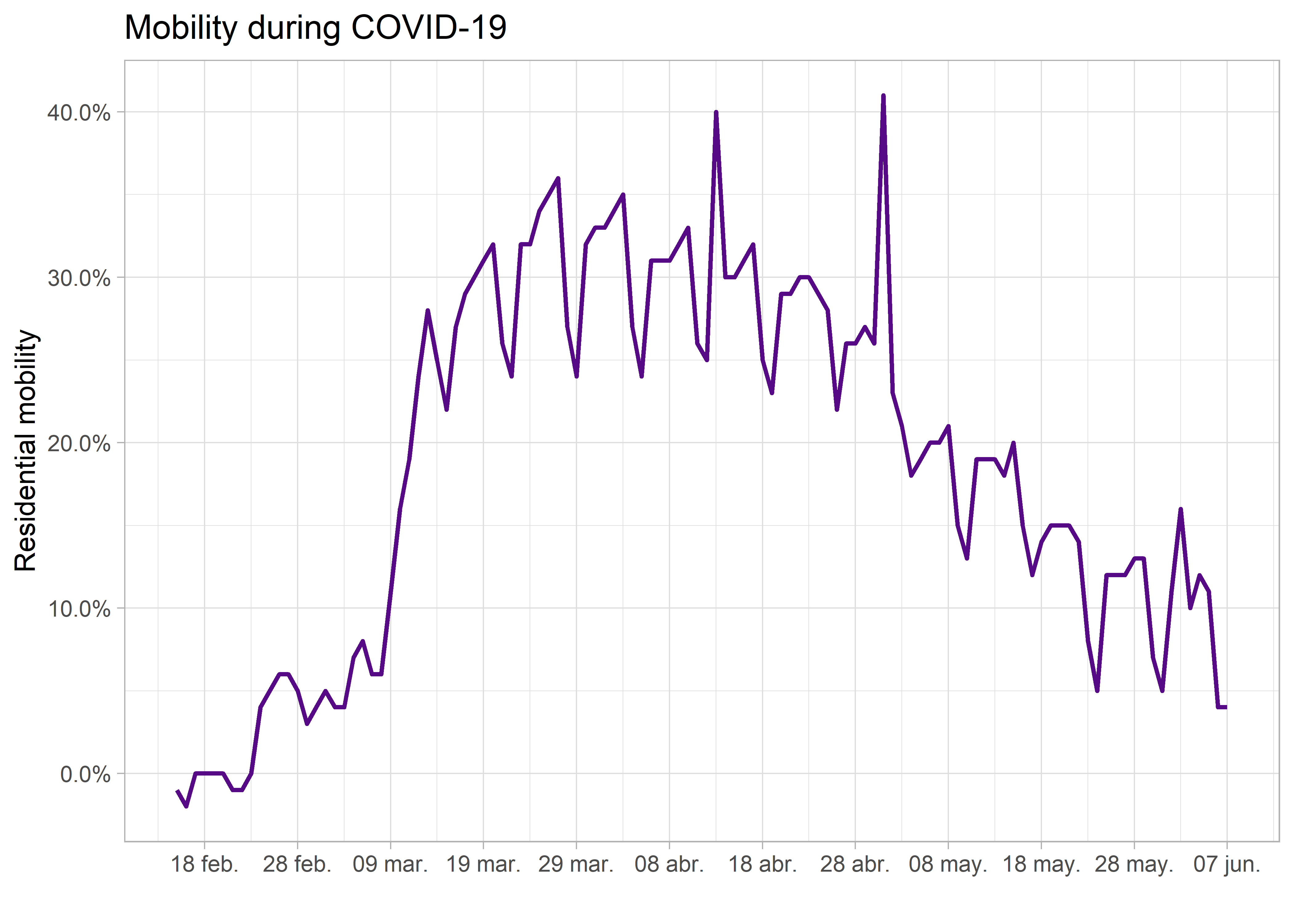
# scatter plot
ggplot(it,
aes(parks, resi)) +
geom_point(alpha = .4, size = 2) +
geom_smooth(method = "lm") +
scale_x_continuous(breaks = seq(-1, 1.4, 0.2),
labels = scales::percent) +
scale_y_continuous(breaks = seq(-1, 1, 0.1),
labels = scales::percent) +
labs(x = "Park mobility",
y = "Residential mobility",
title = "Mobility during COVID-19") +
theme_light()## `geom_smooth()` using formula 'y ~ x'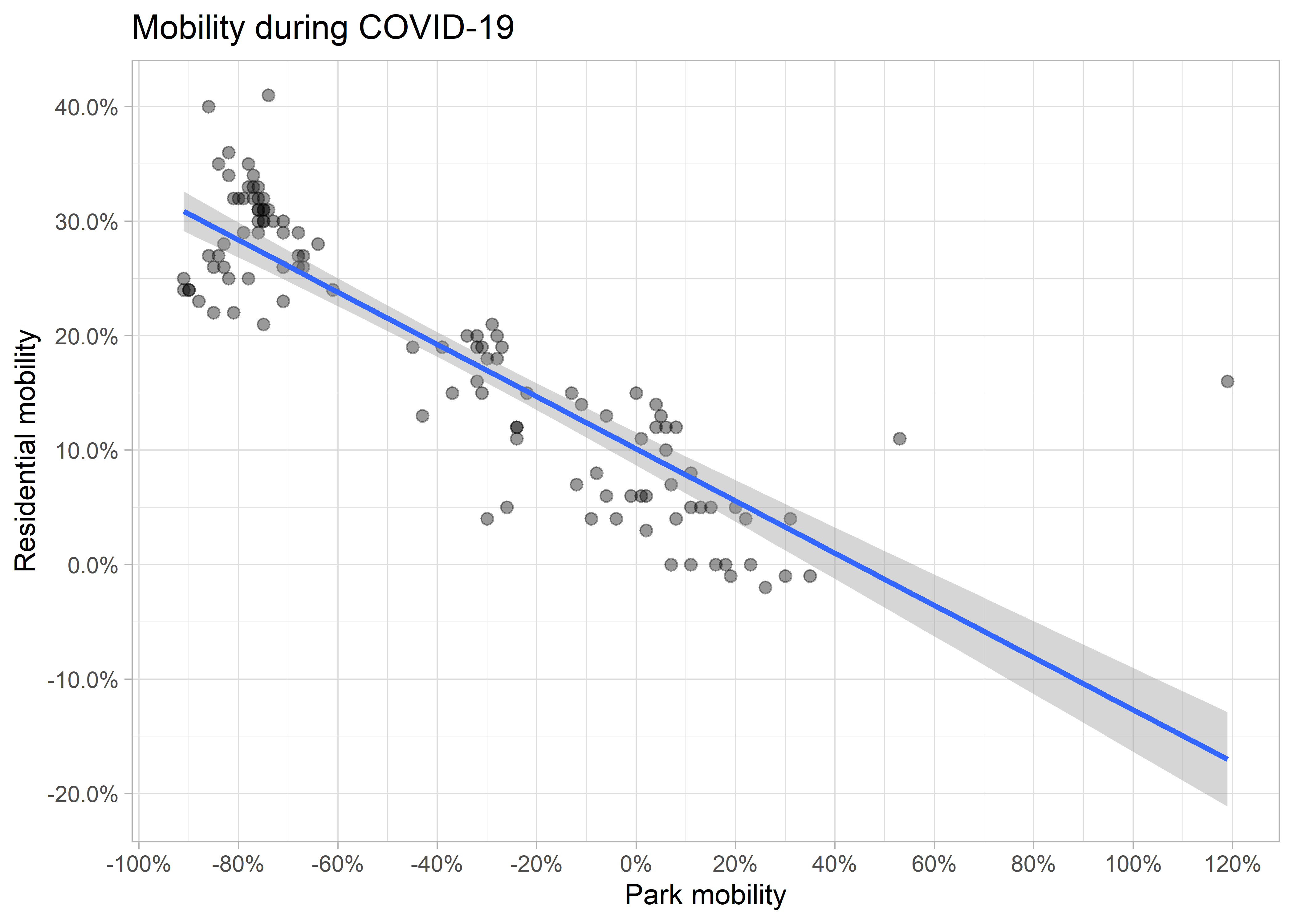
4.5.2 Boxplot
Podemos visualizar diferentes aspectos de los datos de movilidad con otras geometrías. Aquí creamos boxplots por cada país europeo representando la variabilidad de movilidad entre y en los países durante la pandemia del COVID-19.
# subconjunto
subset_europe_reg <- filter(residential_mobility,
!is.na(sub_region_1),
!is.na(resi)) %>%
left_join(wld, by = c("country_region_code"="iso_a2")) %>%
filter(subregion %in% c("Northern Europe",
"Southern Europe",
"Western Europe",
"Eastern Europe")) %>%
mutate(resi = resi/100,
country_region = fct_reorder(country_region, resi))
# boxplot
ggplot(subset_europe_reg,
aes(country_region, resi, fill = subregion)) +
geom_boxplot() +
scale_y_continuous(breaks = seq(-0.1, 1, 0.1), labels = scales::percent) +
scale_fill_brewer(palette = "Set1") +
coord_flip() +
labs(x = "",
y = "Residential mobility",
title = "Mobility during COVID-19",
fill = "") +
theme_minimal()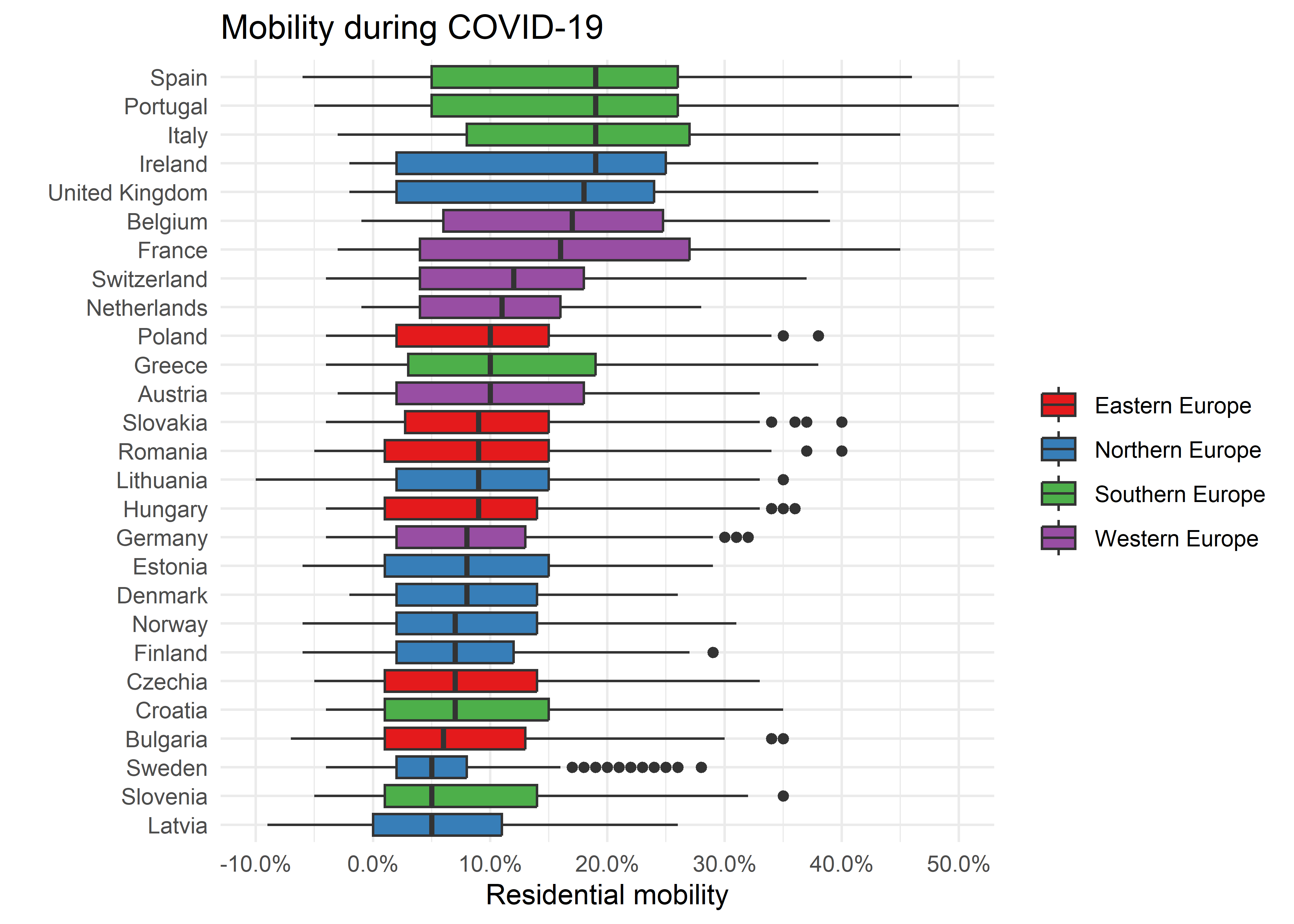
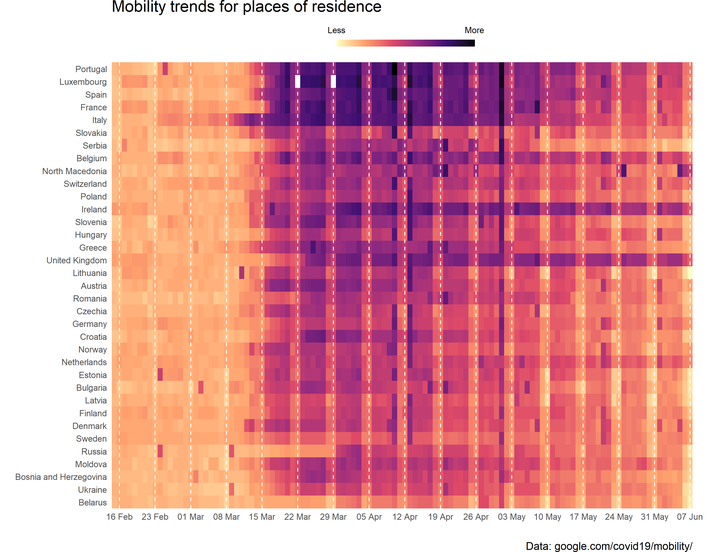
4.5.3 Heatmap
Para visualizar la tendencia de todos los países europeos es recomendable usar un heatmap en lugar de un bulto de líneas. Antes de constuir el gráfico, creamos un vector de fechas para las etiquetas con los domingos en el período de registros.
# secuencia de fechas
df <- data.frame(d = seq(ymd("2020-02-15"), ymd("2020-06-07"), "day"))
# filtramos los domingos creando el día de la semana
sundays <- df %>%
mutate(wd = wday(d, week_start = 1)) %>%
filter(wd == 7) %>%
pull(d)Si queremos usar etiquetas en otras lenguas, es necesario cambiar la configuración regional del sistema.
Sys.setlocale("LC_TIME", "English")## [1] "English_United States.1252"El relleno de color para los boxplots lo dibujamos por cada región de los países europeos. Podemos fijar el tipo de color con scale_fill_*, en este caso, de las gamas viridis.
Además, la función guides() nos permite modificar la barra de color de la leyenda. Por último, aquí vemos el uso de theme() con cambios adicionales a theme_minimal().
# headmap
ggplot(subset_europe,
aes(date, region, fill = resi_real)) +
geom_tile() +
scale_x_date(breaks = sundays,
date_labels = "%d %b") +
scale_fill_viridis_c(option = "A",
breaks = c(91, 146),
labels = c("Less", "More"),
direction = -1) +
theme_minimal() +
theme(legend.position = "top",
title = element_text(size = 14),
panel.grid.major.x = element_line(colour = "white", linetype = "dashed"),
panel.grid.minor.x = element_blank(),
panel.grid.major.y = element_blank(),
panel.ontop = TRUE,
plot.margin = margin(r = 1, unit = "cm")) +
labs(y = "",
x = "",
fill = "",
title = "Mobility trends for places of residence",
caption = "Data: google.com/covid19/mobility/") +
guides(fill = guide_colorbar(barwidth = 10,
barheight = .5,
label.position = "top",
ticks = FALSE)) +
coord_cartesian(expand = FALSE)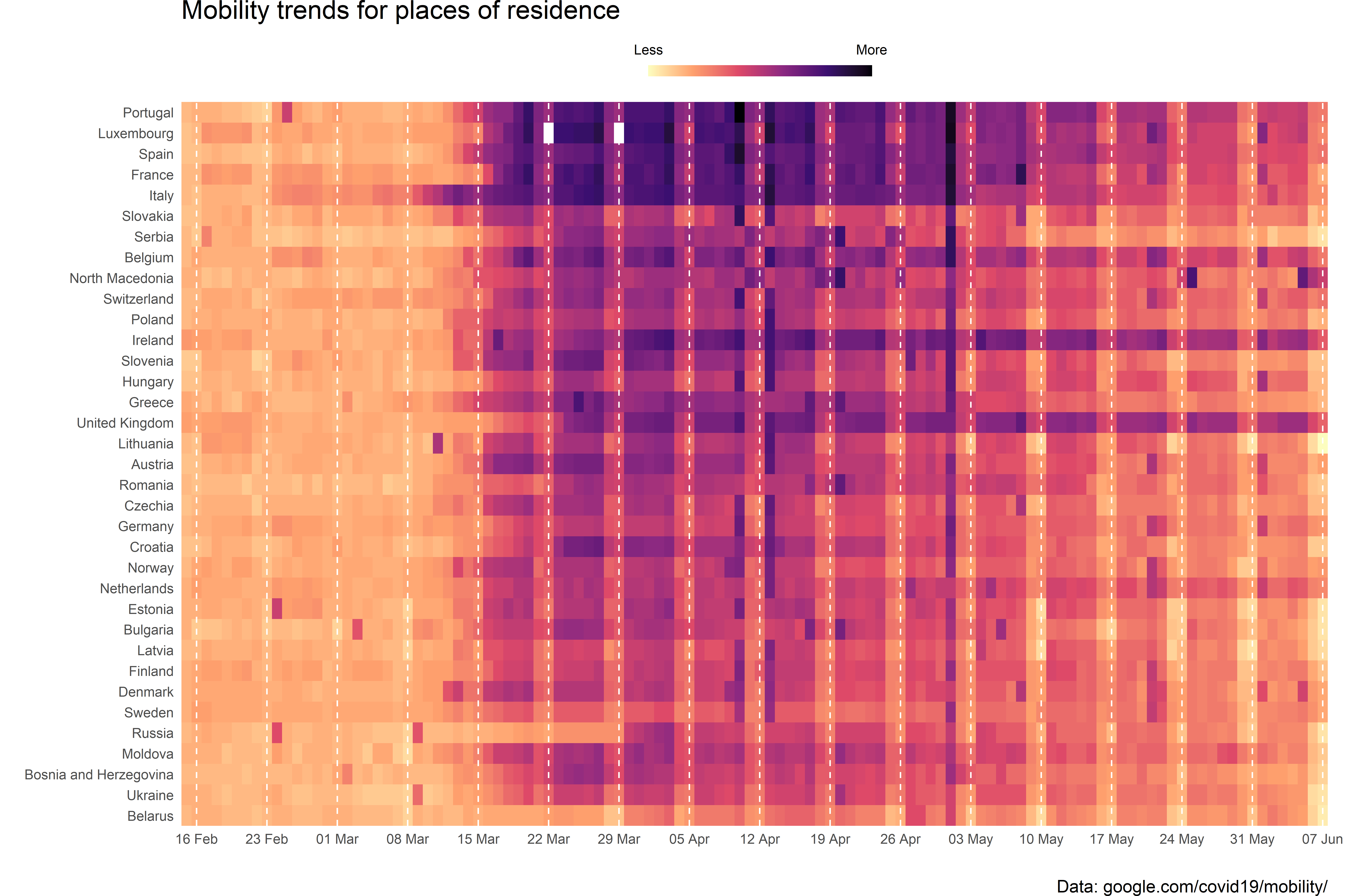
4.6 Aplicar funciones sobre vectores o listas
El paquete purrr contiene un conjunto de funciones avanzadas de programación funcional para trabajar con funciones y vectores. La familia de funciones lapply() conocido de R Basecorresponde a las funciones de map() en este paquete. Una de las mayores ventajas es poder reducir el uso de bucles (for, etc.).
# lista con dos vectores
vec_list <- list(x = 1:10, y = 50:70)
# calculamos el promedio para cada uno
map(vec_list, mean)## $x
## [1] 5.5
##
## $y
## [1] 60# podemos cambiar tipo de salida map_* (dbl, chr, lgl, etc.)
map_dbl(vec_list, mean)## x y
## 5.5 60.0Un ejemplo más complejo. Calculamos el coeficiente de correlación entre la movilidad residencial y la de los parques en todos los países europeos. Para obtener un resumen tidy de un modelo o un test usamos la función tidy() del paquete broom.
library(broom) # tidy outputs
# función adaptada
cor_test <- function(x, formula) {
df <- cor.test(as.formula(formula), data = x) %>% tidy()
return(df)
}
# preparamos los datos
europe_reg <- filter(google_mobility,
!is.na(sub_region_1),
!is.na(residential_percent_change_from_baseline)) %>%
left_join(wld, by = c("country_region_code"="iso_a2")) %>%
filter(subregion %in% c("Northern Europe",
"Southern Europe",
"Western Europe",
"Eastern Europe"))
# aplicamos la función a cada país creando una lista
europe_reg %>%
split(.$country_region_code) %>%
map(cor_test, formula = "~ residential_percent_change_from_baseline + parks_percent_change_from_baseline") ## $AT
## # A tibble: 1 x 8
## estimate statistic p.value parameter conf.low conf.high method alternative
## <dbl> <dbl> <dbl> <int> <dbl> <dbl> <chr> <chr>
## 1 -0.360 -12.3 2.68e-32 1009 -0.413 -0.305 Pearson'~ two.sided
##
## $BE
## # A tibble: 1 x 8
## estimate statistic p.value parameter conf.low conf.high method alternative
## <dbl> <dbl> <dbl> <int> <dbl> <dbl> <chr> <chr>
## 1 -0.312 -6.06 3.67e-9 340 -0.405 -0.213 Pears~ two.sided
##
## $BG
## # A tibble: 1 x 8
## estimate statistic p.value parameter conf.low conf.high method alternative
## <dbl> <dbl> <dbl> <int> <dbl> <dbl> <chr> <chr>
## 1 -0.677 -37.8 1.47e-227 1694 -0.702 -0.650 Pearson~ two.sided
##
## $CH
## # A tibble: 1 x 8
## estimate statistic p.value parameter conf.low conf.high method alternative
## <dbl> <dbl> <dbl> <int> <dbl> <dbl> <chr> <chr>
## 1 -0.0786 -2.91 0.00370 1360 -0.131 -0.0256 Pearson's~ two.sided
##
## $CZ
## # A tibble: 1 x 8
## estimate statistic p.value parameter conf.low conf.high method alternative
## <dbl> <dbl> <dbl> <int> <dbl> <dbl> <chr> <chr>
## 1 -0.0837 -3.35 0.000824 1593 -0.132 -0.0347 Pearson'~ two.sided
##
## $DE
## # A tibble: 1 x 8
## estimate statistic p.value parameter conf.low conf.high method alternative
## <dbl> <dbl> <dbl> <int> <dbl> <dbl> <chr> <chr>
## 1 0.00239 0.102 0.919 1814 -0.0436 0.0484 Pearson's~ two.sided
##
## $DK
## # A tibble: 1 x 8
## estimate statistic p.value parameter conf.low conf.high method alternative
## <dbl> <dbl> <dbl> <int> <dbl> <dbl> <chr> <chr>
## 1 0.237 5.81 1.04e-8 567 0.158 0.313 Pears~ two.sided
##
## $EE
## # A tibble: 1 x 8
## estimate statistic p.value parameter conf.low conf.high method alternative
## <dbl> <dbl> <dbl> <int> <dbl> <dbl> <chr> <chr>
## 1 -0.235 -2.88 0.00462 142 -0.384 -0.0740 Pearson's~ two.sided
##
## $ES
## # A tibble: 1 x 8
## estimate statistic p.value parameter conf.low conf.high method alternative
## <dbl> <dbl> <dbl> <int> <dbl> <dbl> <chr> <chr>
## 1 -0.825 -65.4 0 2005 -0.839 -0.811 Pearson's~ two.sided
##
## $FI
## # A tibble: 1 x 8
## estimate statistic p.value parameter conf.low conf.high method alternative
## <dbl> <dbl> <dbl> <int> <dbl> <dbl> <chr> <chr>
## 1 0.0427 1.42 0.155 1106 -0.0162 0.101 Pearson's~ two.sided
##
## $FR
## # A tibble: 1 x 8
## estimate statistic p.value parameter conf.low conf.high method alternative
## <dbl> <dbl> <dbl> <int> <dbl> <dbl> <chr> <chr>
## 1 -0.698 -37.4 3.29e-216 1474 -0.723 -0.671 Pearson~ two.sided
##
## $GB
## # A tibble: 1 x 8
## estimate statistic p.value parameter conf.low conf.high method alternative
## <dbl> <dbl> <dbl> <int> <dbl> <dbl> <chr> <chr>
## 1 -0.105 -11.0 9.19e-28 10712 -0.124 -0.0865 Pearson'~ two.sided
##
## $GR
## # A tibble: 1 x 8
## estimate statistic p.value parameter conf.low conf.high method alternative
## <dbl> <dbl> <dbl> <int> <dbl> <dbl> <chr> <chr>
## 1 -0.692 -27.0 1.03e-114 796 -0.726 -0.654 Pearson~ two.sided
##
## $HR
## # A tibble: 1 x 8
## estimate statistic p.value parameter conf.low conf.high method alternative
## <dbl> <dbl> <dbl> <int> <dbl> <dbl> <chr> <chr>
## 1 -0.579 -21.9 9.32e-87 954 -0.620 -0.536 Pearson'~ two.sided
##
## $HU
## # A tibble: 1 x 8
## estimate statistic p.value parameter conf.low conf.high method alternative
## <dbl> <dbl> <dbl> <int> <dbl> <dbl> <chr> <chr>
## 1 -0.342 -15.6 6.71e-52 1843 -0.382 -0.301 Pearson'~ two.sided
##
## $IE
## # A tibble: 1 x 8
## estimate statistic p.value parameter conf.low conf.high method alternative
## <dbl> <dbl> <dbl> <int> <dbl> <dbl> <chr> <chr>
## 1 -0.222 -8.45 7.49e-17 1378 -0.271 -0.171 Pearson'~ two.sided
##
## $IT
## # A tibble: 1 x 8
## estimate statistic p.value parameter conf.low conf.high method alternative
## <dbl> <dbl> <dbl> <int> <dbl> <dbl> <chr> <chr>
## 1 -0.831 -71.0 0 2250 -0.844 -0.818 Pearson's~ two.sided
##
## $LT
## # A tibble: 1 x 8
## estimate statistic p.value parameter conf.low conf.high method alternative
## <dbl> <dbl> <dbl> <int> <dbl> <dbl> <chr> <chr>
## 1 -0.204 -5.45 7.17e-8 686 -0.274 -0.131 Pears~ two.sided
##
## $LV
## # A tibble: 1 x 8
## estimate statistic p.value parameter conf.low conf.high method alternative
## <dbl> <dbl> <dbl> <int> <dbl> <dbl> <chr> <chr>
## 1 -0.544 -6.87 3.84e-10 112 -0.662 -0.401 Pearson'~ two.sided
##
## $NL
## # A tibble: 1 x 8
## estimate statistic p.value parameter conf.low conf.high method alternative
## <dbl> <dbl> <dbl> <int> <dbl> <dbl> <chr> <chr>
## 1 0.143 5.31 0.000000125 1356 0.0903 0.195 Pears~ two.sided
##
## $NO
## # A tibble: 1 x 8
## estimate statistic p.value parameter conf.low conf.high method alternative
## <dbl> <dbl> <dbl> <int> <dbl> <dbl> <chr> <chr>
## 1 0.0483 1.69 0.0911 1221 -0.00774 0.104 Pearson's~ two.sided
##
## $PL
## # A tibble: 1 x 8
## estimate statistic p.value parameter conf.low conf.high method alternative
## <dbl> <dbl> <dbl> <int> <dbl> <dbl> <chr> <chr>
## 1 -0.531 -26.7 6.08e-133 1815 -0.564 -0.498 Pearson~ two.sided
##
## $PT
## # A tibble: 1 x 8
## estimate statistic p.value parameter conf.low conf.high method alternative
## <dbl> <dbl> <dbl> <int> <dbl> <dbl> <chr> <chr>
## 1 -0.729 -46.9 2.12e-321 1938 -0.749 -0.707 Pearson~ two.sided
##
## $RO
## # A tibble: 1 x 8
## estimate statistic p.value parameter conf.low conf.high method alternative
## <dbl> <dbl> <dbl> <int> <dbl> <dbl> <chr> <chr>
## 1 -0.640 -56.0 0 4517 -0.657 -0.623 Pearson's~ two.sided
##
## $SE
## # A tibble: 1 x 8
## estimate statistic p.value parameter conf.low conf.high method alternative
## <dbl> <dbl> <dbl> <int> <dbl> <dbl> <chr> <chr>
## 1 0.106 3.93 0.0000909 1367 0.0529 0.158 Pearson~ two.sided
##
## $SI
## # A tibble: 1 x 8
## estimate statistic p.value parameter conf.low conf.high method alternative
## <dbl> <dbl> <dbl> <int> <dbl> <dbl> <chr> <chr>
## 1 -0.627 -11.4 1.98e-23 200 -0.704 -0.535 Pearson'~ two.sided
##
## $SK
## # A tibble: 1 x 8
## estimate statistic p.value parameter conf.low conf.high method alternative
## <dbl> <dbl> <dbl> <int> <dbl> <dbl> <chr> <chr>
## 1 -0.196 -5.70 1.65e-8 810 -0.262 -0.129 Pears~ two.sidedComo ya hemos visto anteriormente, existen subfunciones de map_* para obtener en lugar de una lista un objeto de otra clase, aquí de data.frame.
cor_mobility <- europe_reg %>%
split(.$country_region_code) %>%
map_df(cor_test,
formula = "~ residential_percent_change_from_baseline + parks_percent_change_from_baseline",
.id = "country_code")
arrange(cor_mobility, estimate)## # A tibble: 27 x 9
## country_code estimate statistic p.value parameter conf.low conf.high method
## <chr> <dbl> <dbl> <dbl> <int> <dbl> <dbl> <chr>
## 1 IT -0.831 -71.0 0 2250 -0.844 -0.818 Pears~
## 2 ES -0.825 -65.4 0 2005 -0.839 -0.811 Pears~
## 3 PT -0.729 -46.9 2.12e-321 1938 -0.749 -0.707 Pears~
## 4 FR -0.698 -37.4 3.29e-216 1474 -0.723 -0.671 Pears~
## 5 GR -0.692 -27.0 1.03e-114 796 -0.726 -0.654 Pears~
## 6 BG -0.677 -37.8 1.47e-227 1694 -0.702 -0.650 Pears~
## 7 RO -0.640 -56.0 0 4517 -0.657 -0.623 Pears~
## 8 SI -0.627 -11.4 1.98e- 23 200 -0.704 -0.535 Pears~
## 9 HR -0.579 -21.9 9.32e- 87 954 -0.620 -0.536 Pears~
## 10 LV -0.544 -6.87 3.84e- 10 112 -0.662 -0.401 Pears~
## # ... with 17 more rows, and 1 more variable: alternative <chr>Otros ejemplos prácticos aquí en este post or este otro. Podemos encontrar más detalles en el Cheat-Sheet de purrr.